В самом конце июля, на известном ресурсе bioRxiv наконец-то появился препринт давно ожидаемой статьи тартуских генетиков в составе Баязита Юнусбаева, Майта Метспалу и др., предметом исследования которой является важный вопрос, — оставили ли многочисленные волны экспансии и миграций древних тюрков в структуре генофонда тюркоязычных народов? Следует отметить, что Баязит Юнусбаев и ранее занимался изучением вопроса характера, состава и происхождения генетических компонентов ряда современных тюркских популяций, однако ареал исследований и используемые методы в его предыдущих исследованиях носили ограниченный характер. Свежая работа коллектива тартуских генетиков замечательна уже тем, что в ней было уделено серьезное мнение разработке точного статистико-математического аппарата для определения статистически достоверных геномных cигналов свидетельствующих о определенном характере, направленности и экстенсивности демографических процессов в среде предков современных тюркских популяций.
Наверное, именно по этим причинам работа над подготовкой данных и текста публикации велась довольно долго, не менее 3-4 лет, при том что средний цикл проведений таких исследований на уже готовых генетических данных составляет максимум год-полтора.
Существенным отличием от других подобных работ последнего времени является и заметно явное смещение акцента исследования со ставших уже традционными методов анализа генетических компонентов (кластеров аллельных частот, которые более или менее коррелируют с географией расселения человеческих популяций), таких как PCA, Admixture) на анализ так называемых IBD сегментов и блогов, имеющих общее генетическое происхождение. Как недавно показал на убедительных примерах ув. Сергей Козлов, зачастую правильно распланированный и тщательно выверенный анализ IBD дает более точную, в сравнении с Admixture, генетическую картину происхождения человека. Этот метод основан на метрике IBD и принципиально отличается от Admixture. У него есть свои преимущества, часто он дает лучшую прорисовку кластеров предковых популяций, построенных на основе матрицы разделяемых общих сегментов. Есть и свои недостатки — которые объясняются консервативным характером сохранения некоторых участков. Я бы рекомендовал удалить такие сегменты из анализа — в первую очередь большой мультимаркерный гаплотип региона MHC-HLA на 6 хромосоме, а также ряд участков с высоким уровнем LD. Это значительно улучшит результатЭто наблюдение особенно применимо к относительно гомогенным, однородным популяциям северо-восточной Европы.
Возвращаясь к обсуждаемой статье, можно сказать, что краеугольным рабочим методом в этой статье является расширенный fastIBD анализ большого количества геномных образцов представителей практически всех тюркских народов. В работе присутствуют и более привычные результаты Admixture и PCA анализов структуры генофонда тюркских популяций; однако, на мой личный взгляд, они менее важны в силу тривиальности результатов и легкой повторяемости эксперимента.
Выводы авторов, вынесенные в абстракт статьи, вряд ли вызовут сомнение в своей правильности у большинства историков:
1) Большинство тюркских народов изученых в данной статье, (за исключением тюрков Центральной Азии), генетически напоминают своих географических соседей, что хорошо согласуются с моделью языковой экспансии, в которой тюркские языки — как языки доминирующей элиты -распространялись кочевой элитой.
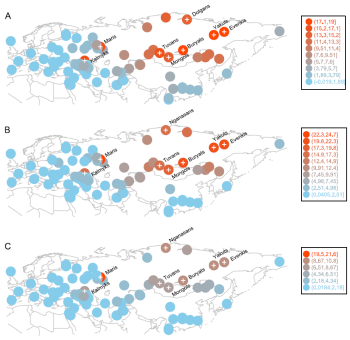
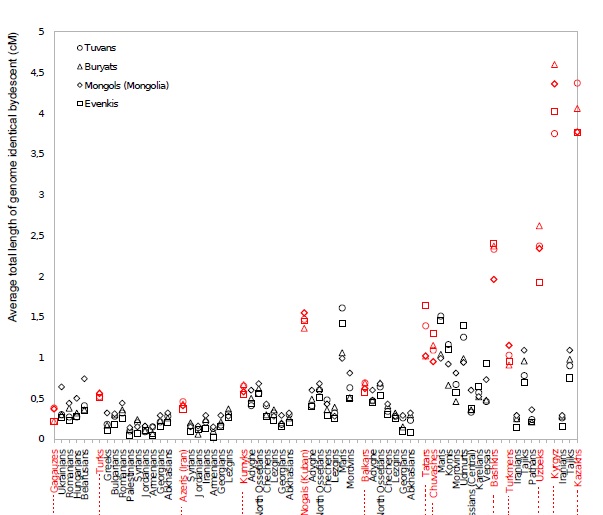
2) 2) Западные тюркские народы в выборке Западной Евразии характеризуются эксцессом длинных хромосомных сегментов, которые идентичны по своему происхождению (IBD) с большей частью населения современной Южной Сибири и Монголии (SSM), т.е в той области, где историки отмечают концетрацию серию ранних тюркских и не -тюркских степных политических объединений. При всем этом, наблюдаемый избыток длинных общих по генеалогическому происхождению IBD сегментов (> 1 сентиморгана) между популяциями из региона Южной Сибирии и Монголии и тюркских народов всей Западной Евразии была статистически значимой.
3) Примененные в исследовании методы датировки событий генетического смешения групп популяций (метод ALDER и SPCO) показали у тюрских народов присутствие сигнала смешивания различных предковых группы в интервале между ~ 9-17-ыми векми нашей эры. Несмотря на принципиальную разницу между этими методами, они дали идентичные результаты, что придает дополнительную надежность вычисленному интервалу И этот интервал перекрывается интервалом тюркских миграций с 5-го по 16 века.
Примечание 1. Мой комментарий
Как я уже отмечал выше, несмотря на всю тривиальность результатов, эту публикацию Юнусбаева et al. 2014 следует отнести к важным работам, поскольку впервые методы оценки времени слияния популяция — ALDER и SPCO были использованы для анализа популяций без явного намека на смешения, расширявшихся в уже историческое время. Ранее эти методы использовались либо при изучении древних доисторических процессов (например, смешивания неолитического и мезолитического населения Европы). либо с использованием классических «смешанных» популяций (мозабитов, пуэрто-риканцев, карибцев и так далее).
В этой связи, заслуживает внимание результаты ALDER для группы тюркских популяций Центральной Азиии (Table 3 в сапплементе к статье), в третьей колонке которой показана датировка событий «смешивания» в поколениях (которые пересчитаны в 4 колонке на года), и это событие приходится на интервал между 13 и 14 веками нашей эры, то есть во времена Золотой Орды :
| Kazakhs | Italians (North Italy) | Tujia | 23.72±1.61 | 1288±48 | 0.00039184±0.00002155 |
| Kyrgyz | Orcadians | Japanese | 22.02±1.00 | 1339±30 | 0.00035833±0.00001271 |
| Uzbeks | Italians (North Italy) | Tujia | 22.07±1.47 | 1338±44 | 0.00036534±0.00001432 |
| Karakalpaks | Italians (North Italy) | Naxi | 22.69±1.89 | 1319±57 | 0.00044112±0.00001912 |
Однако не все просто. Еще в ноябре 2012 года при обсуждении характерных особенностей митохондриальных гаплогрупп жителей Евразии (в статье Клио дер Саркиссян), я решил проверить, насколько эта модель гаплоидной вариативности находит свое подтверждение в анализе диплоидных аутосомных маркеров.
Для этих целей я использовал программу ALDER: Admixture-induced Linkage Disequilibrium for Evolutionary Relationships, специально разработанную для формального обнаружения в анализируемой популяции сигнала смешивания двух и более исходных популяций.В качестве эксперимента я выбрал две современные популяции — казахов и узбеков.
Как видно, полученные мной результаты оказались очень похожи на результаты из более поздней статьи Юнусбаева et al. 2014
Из полученных результатов были отобраны только те успешные результаты, которые прошли формальные критерии отбора (статистический значимый уровень экспонентного угасания неравновесного сцепления маркеров(LD curve is significant) и наличие двухсторонней корреляции между кривыми угасания неравновесного сцепления маркеров в обеих референсных популяциях(decay rates are consistent)).
Результаты по узбекам
DATA: success 3.7e-18 Uzbek Italian-Center Mongol 9.54 9.15 5.18 13% 22.94 +/- 2.41 0.00024041 +/- 0.00001438 23.78 +/- 2.60 0.00006319 +/- 0.00000406 26.14 +/- 5.05 0.00006772 +/- 0.00000894
DATA: success 5.8e-33 Uzbek Sicilian Kyrgyz 12.59 8.51 4.94 19% 23.50 +/- 1.87 0.00015817 +/- 0.00001067 25.77 +/- 3.03 0.00005899 +/- 0.00000443 28.44 +/- 5.76 0.00003069 +/- 0.00000506
DATA: success 6.9e-25 Uzbek Sicilian Mongol 11.03 8.51 5.18 7% 24.49 +/- 2.22 0.00024382 +/- 0.00001210 25.77 +/- 3.03 0.00005899 +/- 0.00000443 26.14 +/- 5.05 0.00006772 +/- 0.00000894
DATA: success 4e-23 Uzbek Sicilian Kalmyk 10.66 8.51 5.56 16% 24.46 +/- 2.29 0.00022326 +/- 0.00001473 25.77 +/- 3.03 0.00005899 +/- 0.00000443 28.67 +/- 5.16 0.00006591 +/- 0.00000891
DATA: success 0.00077 Uzbek Sicilian Nogai 5.12 8.51 2.26 10% 23.79 +/- 4.56 0.00001986 +/- 0.00000388 25.77 +/- 3.03 0.00005899 +/- 0.00000443 23.24 +/- 10.27 0.00001138 +/- 0.00000317
DATA: success 9.8e-21 Uzbek Sardinian Kyrgyz 10.14 9.82 4.94 17% 23.96 +/- 2.36 0.00016455 +/- 0.00001038 27.67 +/- 2.82 0.00007013 +/- 0.00000589 28.44 +/- 5.76 0.00003069 +/- 0.00000506
DATA: success 2e-20 Uzbek Sardinian Mongol 10.07 9.82 5.18 10% 25.15 +/- 2.50 0.00025559 +/- 0.00001310 27.67 +/- 2.82 0.00007013 +/- 0.00000589 26.14 +/- 5.05 0.00006772 +/- 0.00000894
DATA: success 6e-13 Uzbek Sardinian Kalmyk 8.20 9.82 5.56 19% 23.64 +/- 2.88 0.00022058 +/- 0.00001440 27.67 +/- 2.82 0.00007013 +/- 0.00000589 28.67 +/- 5.16 0.00006591 +/- 0.00000891
DATA: success 0.00011 Uzbek Sardinian Nogai 5.48 9.82 2.26 17% 24.99 +/- 4.56 0.00002279 +/- 0.00000367 27.67 +/- 2.82 0.00007013 +/- 0.00000589 23.24 +/- 10.27 0.00001138 +/- 0.00000317
DATA: success 1.5e-28 Uzbek German Kyrgyz 11.77 9.19 4.94 25% 22.14 +/- 1.88 0.00012893 +/- 0.00000925 24.85 +/- 2.70 0.00004544 +/- 0.00000443 28.44 +/- 5.76 0.00003069 +/- 0.00000506
DATA: success 6.9e-21 Uzbek German Mongol 10.17 9.19 5.18 7% 24.40 +/- 2.40 0.00021733 +/- 0.00001182 24.85 +/- 2.70 0.00004544 +/- 0.00000443 26.14 +/- 5.05 0.00006772 +/- 0.00000894
DATA: success 2.8e-16 Uzbek German Kalmyk 9.08 9.19 5.56 22% 23.04 +/- 2.54 0.00018456 +/- 0.00001210 24.85 +/- 2.70 0.00004544 +/- 0.00000443 28.67 +/- 5.16 0.00006591 +/- 0.00000891
Результаты казахов:
DATA: success 4.7e-17 Kazakh Italian-Center Kalmyk 9.27 7.06 2.63 17% 22.06 +/- 2.38 0.00022347 +/- 0.00001893 25.42 +/- 3.60 0.00012981 +/- 0.00001327 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 3.5e-18 Kazakh German Kalmyk 9.54 6.39 2.63 18% 21.71 +/- 2.27 0.00021450 +/- 0.00001602 23.54 +/- 3.68 0.00012169 +/- 0.00001026 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 2.6e-23 Kazakh Russian_Center Kalmyk 10.70 6.64 2.63 17% 22.19 +/- 2.07 0.00023388 +/- 0.00001645 21.86 +/- 3.29 0.00012520 +/- 0.00001320 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 2.1e-22 Kazakh Russian_South Kalmyk 10.50 7.12 2.63 25% 20.31 +/- 1.93 0.00021745 +/- 0.00001580 20.82 +/- 2.93 0.00012386 +/- 0.00001116 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 0.019 Kazakh Slovakian Mari 4.48 8.09 3.45 17% 17.26 +/- 3.86 0.00002773 +/- 0.00000574 19.08 +/- 2.36 0.00011870 +/- 0.00001088 16.06 +/- 4.65 0.00003481 +/- 0.00000667
DATA: success 1.6e-29 Kazakh Ukrainian Kalmyk 11.95 6.93 2.63 23% 20.58 +/- 1.41 0.00021665 +/- 0.00001813 20.75 +/- 3.00 0.00011940 +/- 0.00001005 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 1.5e-14 Kazakh Ukrainian-East Kalmyk 8.63 5.90 2.63 23% 20.58 +/- 2.38 0.00022215 +/- 0.00001803 21.97 +/- 3.72 0.00012517 +/- 0.00001419 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 0.0014 Kazakh CEU_V Nogai 5.02 5.49 3.30 17% 20.84 +/- 4.16 0.00001984 +/- 0.00000315 19.20 +/- 3.50 0.00012065 +/- 0.00001375 17.52 +/- 5.31 0.00004319 +/- 0.00000772
DATA: success 0.00025 Kazakh British Mari 5.33 6.99 3.45 24% 20.42 +/- 3.83 0.00003281 +/- 0.00000478 19.18 +/- 2.74 0.00012196 +/- 0.00001159 16.06 +/- 4.65 0.00003481 +/- 0.00000667
DATA: success 0.0064 Kazakh British Nogai 4.71 6.99 3.30 18% 21.09 +/- 4.48 0.00002087 +/- 0.00000321 19.18 +/- 2.74 0.00012196 +/- 0.00001159 17.52 +/- 5.31 0.00004319 +/- 0.00000772
DATA: success 5.8e-22 Kazakh Orcadian Kalmyk 10.41 6.62 2.63 23% 20.59 +/- 1.98 0.00023474 +/- 0.00001737 21.83 +/- 3.30 0.00013779 +/- 0.00001201 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 0.004 Kazakh Bulgarian Nogai 4.80 6.98 3.30 21% 21.66 +/- 4.51 0.00001853 +/- 0.00000339 21.33 +/- 3.06 0.00012336 +/- 0.00001168 17.52 +/- 5.31 0.00004319 +/- 0.00000772
DATA: success 3.1e-19 Kazakh Karelian Kalmyk 9.79 5.77 2.63 21% 21.05 +/- 2.15 0.00019192 +/- 0.00001302 21.12 +/- 3.66 0.00009774 +/- 0.00001073 26.05 +/- 8.19 0.00002219 +/- 0.00000844
DATA: success 0.011 Kazakh Mari Bosnian 4.60 3.45 6.44 16% 17.56 +/- 3.82 0.00003003 +/- 0.00000554 16.06 +/- 4.65 0.00003481 +/- 0.00000667 18.79 +/- 2.92 0.00012259 +/- 0.00001136
DATA: success 0.0057 Kazakh Mari Greek_Azov 4.73 3.45 9.00 21% 16.97 +/- 3.59 0.00002322 +/- 0.00000431 16.06 +/- 4.65 0.00003481 +/- 0.00000667 19.87 +/- 2.21 0.00010948 +/- 0.00000811
DATA: success 5.2e-33 Kazakh Chuvash Kalmyk 12.60 5.88 2.63 14% 24.10 +/- 1.91 0.00014440 +/- 0.00000896 22.75 +/- 3.87 0.00005482 +/- 0.00000595 26.05 +/- 8.19 0.00002219 +/- 0.00000844
Результаты говорят сами за себя. Как и в большинстве случаев с центральноазиатскими популяциями, один из компонентов адмикса у узбекв и казахов представлен монголоидным популяциями, наиболее близкими к современным монголам, киргизам и калмыкам. В контексте обсуждаемой работы о древнем митоДНК, этот компонент можно обозначать как «восточноевразийский». Другой компонент у узбеков представлен популяциями близкими по частотам аллелей к современным западноевропейским популяциям (таким как немцы, cардинцы, сицилийцы и прочие). Кроме того в результатах анализа угасания admixture-LD казахов присутствует хорошо заметный сигнал адмикса с предками современных чувашей, марийцев и карел. Этот феномен, опять-таки в контексте парадигмы исследования древнего ДНК, можно объяснить с помощью грубой аппроксимации: как было убедительно показано в работе Дерсаркиссян, митоДНК древние скифы из ареала современной Кубани и нижнего Поволжья напоминают ряд современных поволжских популяций, а также карелов. Это же касается и сигнала о смешивании с предками более отдаленных европейских популяций (британцев, скандинавов и так далее). В свете аутосомного анализа вынужден согласится с одним из ключевых выводов Дерсаркиссян, согласно которому западноевропейские аллели были привнесены в степени Казахстана и Алтай при посредничестве популяций скифов и сарматов.
Стоит еще отметить, что у узбеков в отличие от казахов поток европейских аллелей имеет несколько иной источник, и скорее всего связан с распространением в Центральной Азии носителей «неолитических средиземнорских аллелей».
Подводя итоги, необходимо сказачть, что определенные трудности представляет интерпретация датировки европейского адмикса у казахов и узбеков. Поскольку этот адмикс датируется примерно одинаковым интервалом 30-25 поколений до нашего времени, что примерно соответсвует периоду завоевательных походов монголов. Однако, представляется трудно допустимым, что европеидное население казахской степи могло сохранится в значительном количестве вплоть до эпохи монголов, или что земледельческое население Средней Азии — сарты — было в генетическом плане европеоидно. Лично я склоняюсь к следущей интерпретации: смешивание европейского и восточноазиатского компонента произошло намного ранее монгольской эпохи, заниженная дата этого события есть прямое следствие последущего эфекта дрейфа генов и фиксации части аллелей.
Примечание 2. Комментарий профессионального историка
Хорошая работа. Но это — еще самое начало пути. Пока идет процесс верификации используемых методов. Очень важно, что результаты в целом совпали с тем, что говорят исторические источники. Для генетиков это хорошо, но для историков — тривиально. Действительный интерес появится, когда станет возможно давать такие ответы, которые историки своими методами добыть не могут. А этого пока нет.
Я на заре своей научной карьеры аналогичные выводы получил, используя всего один интегрированный фенотипический признак — индекс уплощенности лицевого скелета. По трудозатратам это было несопоставимо. Да, теперь эти выводы обоснованы гораздо надежнее. Но хочется гораздо большего.
Генетические следы экспансии тюркоязычных номадов в Евразии: Один комментарий