После нескольких лет практически полного пассивного бездействия в области изучения генетической вариативности популяций населения Восточной Европы, я решил продолжить свои скромные изыскания в этом вопросе. Примерно год ушел на пересборку и соединение различных выборок популяций (выборки разных исследований содержат разное количество частично перекрывающихся снипов, и это обстоятельство существенно влияет на качество и значимость получаемыых в анализе таких выборок результатов). К сожалению, разница в частотах минорных снипов в выборках одних и тех же этнических групп, но генотипированных на разных платформах Illumina и Affy, приводит к существенному снижению качества импутирования недостающих маркеров. Это очень плохо, так как во многих из разработанных методик анализа генетического разнообразия, надежность результатов напрямую зависит от полноты генотипирования, т.е. в идеале во всех популяциях должны быть равномерно представлены все снипы, т.е маркеры из полного объединенного набора. Вопреки моим ожиданиям, у этой проблемы не существует тривиального решения, поэтому я решил отложить задачу импутации отсутствующих генотипов в образцах выборки на дальнейшее (чуть позже я поделюсь своими соображениями о том, как сделать результат импутирования более точным).
Ровно год назад я сообщил о том, что в основу (базу) новой выборки будут положены полный публичный кураторский набор контрольных популяционных групп лаборатории Райха, что и было выполнено частично, хотя запланированную процедуру импутирования так и не удалось завершить в силу огромной компьютерной ресурсозатратности задачи.
Все же, с учетом тяжелых уроков всех предыдущих ошибок (в том числе и при работе с палеогеномами человека), мне все же удалось собрать набор из примерно 6500 сэмплов из более чем 250 этно-популяционных групп со всего земного шара. На этом можно считать первую фазу законченной.
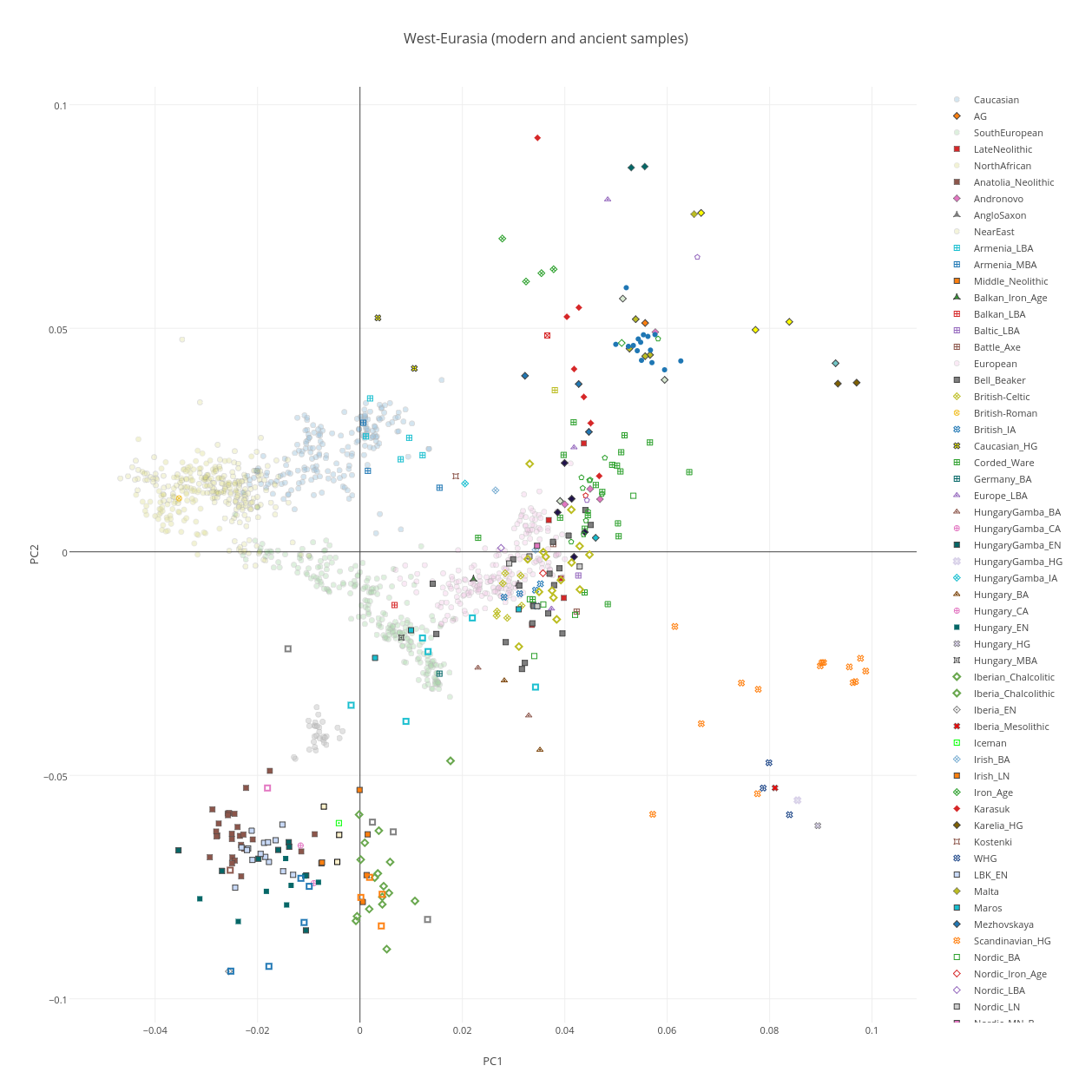
В качестве предварительной иллюстрации надежности результатов можно привести график PCA (анализа главных компонентов генетической вариативности в западноевразийских популяциях из описанной выше сводной выборки, после применения соответствующих фильтров контроля качества снипов).
Как мне кажется, получился неплохой график PC (анализа главных компонент) древних и современных групп народонаселения, причем хорошо видно на какие современные группы накладываются палеогеномы.
Но теперь другая проблема — я не могу сохранить этот график в формате PDF (видимо, разработчики Plotly отключили эту опцию в бесплатной версии). Можно выгрузить графику в файл png, но в отличие от векторного формата pdf, png — формат растровый, и улучшить качество графики уже не получится.
Поэтому я сделал альтернативные варианты (без использования пакета Plotly) графика с изображением положения популяций в пространстве двух главных компонентов генетического разнообразия Евразии.
После несколько лет практически полного отсутствия активности в области изучения генетической вариативности популяций населения Восточной Европы, я решил продолжить свои скромные изыскания в этом вопросе. Примерно год ушел на пересборку и соединение различных выборок популяций (выборки разных исследований содержат разное количество частично перекрывающихся снипов, и это обстоятельство существенно влияет на статистическое качество). К сожалению, разница в частотах минорных снипов в выборках одних и тех же этнических групп, но генотипированных на разных платформах Illumina и Affy, приводит к существенному снижению импутирования недостающих маркеров. Это очень плохо, так как во многих из предложенных методик анализа генетического разнообразия, надежность результатов напрямую зависит от полноты генотипирования, т.е. в идеале во всех популяциях должны быть равномерно представлены все снипы, т.е маркеры из полного объединенного набора. Вопреки моим ожиданиям, у этой проблемы не существует тривиального решения, поэтому я решил отложить задачу импутации отсутствующих генотипов в образцах выборки на дальнейшее (чуть позже я поделюсь своими соображениями о том, как сделать результат импутирования более точным).
Ровно год назад я сообщил о том, что в основу (базу) новой выборки будут положены полный публичный кураторский набор контрольных популяционных групп лаборатории Райха, что и было выполнено частично, хотя запланированную процедуру импутирования так и не удалось завершить в силу огромной компьютерной ресурсозатратности задачи.
Все же, с учетом тяжелых уроков всех предыдущих ошибок (в том числе и при работе с палеогеномами человека), мне все же удалось собрать набор из примерно 6500 сэмплов из более чем 250 этно-популяционных групп со всего земного шара. На этом можно считать первую фазу законченной.
В качестве предварительной иллюстрации надежности результатов можно привести график PCA (анализа главных компонентов генетической вариативности в западноевразийских популяциях из описанной выше сводной выборки, после применения соответствующих фильтров контроля качества снипов).
Как мне кажется, получился неплохой график PC (анализа главных компонент) древних и современных групп народонаселения, причем хорошо видно на какие современные группы накладываются палеогеномы.
Но теперь другая проблема — я не могу сохранить этот график в формате PDF (видимо, разработчики Plotly отключили эту опцию в бесплатной версии). Можно выгрузить графику в файл png, но в отличие от векторного формата pdf, png — формат растровый, и улучшить качество графики уже не получится.
Поэтому я сделал альтернативные варианты (без использования пакета Plotly) графика с изображением положения популяций в пространстве двух главных компонентов генетического разнообразия Евразии.