В качестве одного из метода проверки надежности импутированных снипов для анализа популяционной истории различных этнических групп, я использовал метод ALDER (он представляет собой более продвинутую версию алгоритма ROLOFF, описанного в известной работе Patterson et al. 2012).
Метод ALDER выявляет нюансы популяционной истории через оценку двух важных параметров: а) рекомбинации — процесса обмена участками между разными молекулами ДНК, который напоминает перемешивание игральных карт в колоде (у человека он обязательно происходит при образовании половых клеток) и б) неравновесия по сцеплению — явления, при котором несколько участков ДНК передаются вместе блоками, которые формируются несколько по-разному в разных популяциях из-за того, что в разных популяциях наследуются разные комбинации сегментов ДНК. Таким образом, метод основан на выявлении специфических для каждой популяции сцепленных участков ДНК и на оценке доли общих сегментов в выборках сравниваемых популяций. При этом метод ALDER на основе оценки неравновесия по сцеплению определяет правдоподобность того, что две выбранные группы являются предковыми по отношению к анализируемым популяциям. Кроме того, метод позволяет также установить время смешения через оценку доли рекомбинаций на поколение.
Как было сказано выше, метод ALDER представляет собой расширенный вариант алгоритма ROLLOFF.Этот метод позволяет производить математическую оценку как времени, так как и уровня адмикса. Оценка производится на основании анализа неравновесия по сцеплению между SNP-ами. Тут необходимо вспомнить стандартное определение неравновесия по сцеплению.Неравновесием по сцеплению (часто используется английская аббревиатура LD) называется неслучайная связь между двумя аллелями, в силу которой определенные комбинации аллелей встречаются наиболее часть. В теории, чем дальше друг от друга находятся SNP-ы ,тем меньше будет уровень LD. Темп угасания снижения LD в адмиксе напрямую связана с числом поколений, прошедших с момента адмикса, так как c возрастанием числа поколений увлечивается число рекомбинаций произошедших между двумя отдельными SNP-ами. Проще говоря: Rolloff соответствует экспоненциальной кривой угасания уровня LD от расстояния, и эта скорость экспоненциального снижения как раз и используется для оценки числа поколений, так и уровня адмикса в анализируемой популяии. Учитывая, что одно поколение примерно равно 29 лет, можно преобразовать число поколений в года.В качестве двух тестовых популяций я использовал две группы — выборку белорусов (данные публичной коллекции эстонского биоцентра, вошедшие позднее в стандартный набор популяций лаборатории Райха, а также данные белорусов, собранные мной в проекте MDLP) и выборку поляков (из публичной коллекции эстонского биоцентра, а также данные поляков из выборки моего проекта MDLP). В 2012 году я уже проводил анализ ROLLOFF белорусов, поэтому было интересно посмотреть, как изменится картина после добавления новых палеогеномов и импутированных (негенотипированных) снипов. Для полноты эксперимента, я также включил данные поляков, чтобы посмотреть, работает ли метод на данных, полученных с помощью других платформ генотипирования (NB:когда я проводил анализ, у меня еще не было данных поляков из кураторской базы данных лаборатории Reich).
В качественных референсных популяций (кандидатов в предковые популяции) я использовал набор из 400 популяций в моей расширенной выборки. Программа работает в три этапа:
- На первом этапе определяется статистическая значимость сравнения амплитуд угасания 2-референсной LD(в случае наличия такой значимость программа пишет success)
Cледующие комбинации дали положительный результат
| Belarusian | Andronovo.SG | Chukchis |
| Belarusian | Andronovo.SG | Koryaks |
| Belarusian | Armenian_Martuni | Karelia_HG |
| Belarusian | Bashkir | Turkish_Jewish |
| Belarusian | Nordic_LN.SG | Villabruna |
| Belarusian | Turkish_Jewish | Uzbek |
| Belarusian | Anatolia_Neolithic | Brahui |
| Belarusian | Anatolia_Neolithic | Burusho |
| Belarusian | Anatolia_Neolithic | Itelmen |
| Belarusian | Anatolia_Neolithic | Koryak |
| Belarusian | Anatolia_Neolithic | Mixtec |
| Belarusian | Anatolia_Neolithic | Pathan |
| Belarusian | Anatolia_Neolithic | Mala |
| Belarusian | Anatolia_Neolithic | Turkmen |
| Belarusian | Anatolia_Neolithic | Uygur |
| Belarusian | Druze | Selkup |
| Belarusian | Mala | Syrian |
| Belarusian | Mixtec | Spain_EN |
| Belarusian | Anatolia_Neolithic | Brahui |
| Belarusian | Anatolia_Neolithic | Burusho |
| Belarusian | Anatolia_Neolithic | Chukchi |
| Belarusian | Anatolia_Neolithic | Selkup |
| Belarusian | Anatolia_Neolithic | Sindhi |
| Belarusian | Anatolia_Neolithic | Uygur |
| Belarusian | British-Roman | Koryak |
| Belarusian | British-Roman | Mixtec |
| Belarusian | Chukchi | Mala |
| Belarusian | Itelmen | Uzbek_WGA |
| Belarusian | LBK_EN | Selkup |
| Belarusian | Selkup | Turkish_Trabzon |
| Belarusian | Abhkasian | Lahu |
| Belarusian | Ami_Coriell | Uzbek_WGA |
| Belarusian | Anatolia_Neolithic | Chukchi |
| Belarusian | Anatolia_Neolithic | Daur |
| Anatolia_Neolithic | Han | |
| Anatolia_Neolithic | Han_NChina | |
| Anatolia_Neolithic | Miao | |
| Anatolia_Neolithic | Turkmen | |
| Belarusian | Atayal_Coriell | Uzbek_WGA |
| Belarusian | British-Roman | Mixtec |
| Belarusian | Chukchi | Mala |
| Belarusian | Dai | Greek_Islands |
| Belarusian | Dai | Uzbek_WGA |
| Belarusian | Daur | North_Ossetian |
| Belarusian | Daur | Uzbek_WGA |
| Belarusian | Eskimo_Chaplin | LBK_EN |
| Belarusian | Georgian | Lahu |
| Belarusian | Georgian | Yi |
| Belarusian | Greek_Islands | Han |
| Belarusian | Greek_Islands | Miao |
| Belarusian | Greek_Islands | Mixtec |
| Belarusian | Greek_Islands | Nganasan |
| Belarusian | Greek_Islands | Ulchi |
| Belarusian | Greek_Islands | Xibo |
| Belarusian | Han | Uzbek_WGA |
| Belarusian | Han | Yemenite_Jew |
| Belarusian | Han_NChina | Uzbek_WGA |
| Belarusian | Han_NChina | Yemenite_Jew |
| Belarusian | Japanese | Uzbek_WGA |
| Belarusian | Korean | Uzbek_WGA |
| Belarusian | Lahu | Turkish_Jew |
| Belarusian | Lahu | Uzbek_WGA |
| Belarusian | Lahu | Yemenite_Jew |
| Belarusian | LBK_EN | Selkup |
| Belarusian | Miao | Uzbek_WGA |
| Belarusian | Miao | Yemenite_Jew |
| Belarusian | Naxi | Uzbek_WGA |
| Belarusian | Oroqen | Uzbek_WGA |
| Belarusian | She | Uzbek_WGA |
| Belarusian | Tu | Uzbek_WGA |
| Belarusian | Tujia | Uzbek_WGA |
| Belarusian | Tujia | Yemenite_Jew |
| Belarusian | Ulchi | Uzbek_WGA |
| Belarusian | Uzbek_WGA | Xibo |
| Belarusian | Uzbek_WGA | Yi |
| Belarusian | Uzbek_WGA | Yukagir_Tundra |
| Belarusian | Yemenite_Jew | Yi |
- На втором — cоответствие скоростей угасания LD в попарном сравнении с референсными популяциями (программа выдает предупреждение, если амплитуды угасания LD несовместимы). Как видно, большинство триплетов (таргетная популяция + 2 референса) имеет несовместимые амплитуды угасания LD.
DATA: success (warning: decay rates inconsistent) 0.028 Belarusian Andronovo.SG Chukchis 4.64 2.80 2.11 85% 244.96 +/- 44.45 0.00055485 +/- 0.00011964 262.22 +/- 50.30 0.00029724 +/- 0.00010632 105.99 +/- 50.22 0.00013405 +/- 0.00003707
DATA: success (warning: decay rates inconsistent) 3.8e-05 Belarusian Andronovo.SG Koryaks 5.86 2.80 2.36 85% 241.36 +/- 36.30 0.00059837 +/- 0.00010219 262.22 +/- 50.30 0.00029724 +/- 0.00010632 105.75 +/- 44.80 0.00011083 +/- 0.00002791
DATA: success (warning: decay rates inconsistent) 0.037 Belarusian Armenian_Martuni Karelia_HG 4.58 2.20 3.48 53% 206.14 +/- 39.11 0.00072944 +/- 0.00015918 324.91 +/- 90.64 0.00018302 +/- 0.00008311 189.01 +/- 42.42 0.00043186 +/- 0.00012423
DATA: success (warning: decay rates inconsistent) 0.044 Belarusian Bashkir Turkish_Jewish 4.55 2.70 2.53 83% 121.78 +/- 24.93 0.00009384 +/- 0.00002064 153.64 +/- 48.19 0.00006384 +/- 0.00002366 296.25 +/- 73.05 0.00014988 +/- 0.00005929
DATA: success (warning: decay rates inconsistent) 0.045 Belarusian Nordic_LN.SG Villabruna 4.54 2.19 5.01 30% 160.01 +/- 35.24 0.00086280 +/- 0.00018037 139.86 +/- 63.88 0.00033916 +/- 0.00014340 117.88 +/- 23.51 0.00043952 +/- 0.00008295
DATA: success (warning: decay rates inconsistent) 0.0032 Belarusian Turkish_Jewish Uzbek 5.07 2.53 2.35 112% 116.58 +/- 23.00 0.00008493 +/- 0.00001382 296.25 +/- 73.05 0.00014988 +/- 0.00005929 83.46 +/- 35.51 0.00004275 +/- 0.00001721
DATA: success (warning: decay rates inconsistent) 0.0066 Belarusian Anatolia_Neolithic Brahui 4.18 4.42 2.87 37% 63.78 +/- 15.17 0.00000803 +/- 0.00000192 92.46 +/- 19.49 0.00001427 +/- 0.00000323 88.94 +/- 27.82 0.00000775 +/- 0.00000270
DATA: success (warning: decay rates inconsistent) 0.019 Belarusian Anatolia_Neolithic Burusho 3.93 4.42 2.19 47% 93.43 +/- 9.05 0.00001536 +/- 0.00000390 92.46 +/- 19.49 0.00001427 +/- 0.00000323 149.25 +/- 37.02 0.00001357 +/- 0.00000621
DATA: success (warning: decay rates inconsistent) 0.035 Belarusian Anatolia_Neolithic Itelmen 3.79 4.42 2.15 64% 69.11 +/- 15.92 0.00002889 +/- 0.00000762 92.46 +/- 19.49 0.00001427 +/- 0.00000323 134.23 +/- 58.31 0.00003278 +/- 0.00001523
DATA: success (warning: decay rates inconsistent) 0.023 Belarusian Anatolia_Neolithic Koryak 3.90 4.42 2.30 30% 82.94 +/- 21.28 0.00003363 +/- 0.00000828 92.46 +/- 19.49 0.00001427 +/- 0.00000323 111.83 +/- 48.56 0.00002562 +/- 0.00000985
DATA: success 0.014 Belarusian Anatolia_Neolithic Mala 4.01 4.42 3.87 12% 81.90 +/- 17.24 0.00001547 +/- 0.00000385 92.46 +/- 19.49 0.00001427 +/- 0.00000323 87.55 +/- 18.75 0.00001071 +/- 0.00000277
DATA: success (warning: decay rates inconsistent) 0.025 Belarusian Anatolia_Neolithic Mixtec 3.87 4.42 2.73 71% 71.36 +/- 14.69 0.00003027 +/- 0.00000782 92.46 +/- 19.49 0.00001427 +/- 0.00000323 149.36 +/- 43.83 0.00002944 +/- 0.00001080
DATA: success (warning: decay rates inconsistent) 0.019 Belarusian Anatolia_Neolithic Pathan 3.93 4.42 2.02 42% 104.78 +/- 14.08 0.00001497 +/- 0.00000380 92.46 +/- 19.49 0.00001427 +/- 0.00000323 141.03 +/- 36.42 0.00001165 +/- 0.00000577
DATA: success 0.00024 Belarusian Anatolia_Neolithic Turkmen 4.88 4.42 2.26 12% 81.66 +/- 10.18 0.00001288 +/- 0.00000264 92.46 +/- 19.49 0.00001427 +/- 0.00000323 85.64 +/- 28.96 0.00000855 +/- 0.00000378
DATA: success (warning: decay rates inconsistent) 0.026 Belarusian Anatolia_Neolithic Uygur 3.87 4.42 2.54 56% 71.95 +/- 14.95 0.00001528 +/- 0.00000395 92.46 +/- 19.49 0.00001427 +/- 0.00000323 127.39 +/- 37.67 0.00001541 +/- 0.00000606
DATA: success (warning: decay rates inconsistent) 0.02 Belarusian Druze Selkup 3.93 2.02 3.14 73% 51.53 +/- 13.06 0.00001224 +/- 0.00000311 110.46 +/- 43.38 0.00001040 +/- 0.00000516 59.53 +/- 18.98 0.00000945 +/- 0.00000299
DATA: success (warning: decay rates inconsistent) 0.044 Belarusian Mala Syrian 3.73 3.87 2.84 28% 72.39 +/- 19.33 0.00000805 +/- 0.00000216 87.55 +/- 18.75 0.00001071 +/- 0.00000277 96.31 +/- 27.52 0.00000993 +/- 0.00000350
DATA: success (warning: decay rates inconsistent) 0.027 Belarusian Mixtec Spain_EN 3.85 2.73 2.67 26% 114.65 +/- 21.37 0.00005462 +/- 0.00001417 149.36 +/- 43.83 0.00002944 +/- 0.00001080 117.07 +/- 30.31 0.00002193 +/- 0.00000820
DATA: success (warning: decay rates inconsistent) 0.045 Belarusian Anatolia_Neolithic Brahui 3.70 3.45 2.55 32% 63.07 +/- 15.18 0.00000871 +/- 0.00000235 81.39 +/- 21.48 0.00001207 +/- 0.00000349 87.19 +/- 27.66 0.00000771 +/- 0.00000303
DATA: success (warning: decay rates inconsistent) 0.039 Belarusian Anatolia_Neolithic Burusho 3.74 3.45 2.00 61% 89.47 +/- 10.12 0.00001582 +/- 0.00000423 81.39 +/- 21.48 0.00001207 +/- 0.00000349 152.62 +/- 45.80 0.00001482 +/- 0.00000742
DATA: success (warning: decay rates inconsistent) 0.0013 Belarusian Anatolia_Neolithic Chukchi 4.52 3.45 2.79 35% 77.64 +/- 16.37 0.00003602 +/- 0.00000797 81.39 +/- 21.48 0.00001207 +/- 0.00000349 110.36 +/- 39.54 0.00002861 +/- 0.00000981
DATA: success (warning: decay rates inconsistent) 0.038 Belarusian Anatolia_Neolithic Selkup 3.74 3.45 2.41 38% 55.27 +/- 13.63 0.00002155 +/- 0.00000576 81.39 +/- 21.48 0.00001207 +/- 0.00000349 57.06 +/- 20.02 0.00000933 +/- 0.00000386
DATA: success (warning: decay rates inconsistent) 0.027 Belarusian Anatolia_Neolithic Sindhi 3.83 3.45 2.68 61% 65.40 +/- 9.12 0.00001072 +/- 0.00000280 81.39 +/- 21.48 0.00001207 +/- 0.00000349 122.70 +/- 32.60 0.00001132 +/- 0.00000423
DATA: success 0.00044 Belarusian Anatolia_Neolithic Turkmen 4.74 3.45 3.07 11% 76.01 +/- 10.03 0.00001185 +/- 0.00000250 81.39 +/- 21.48 0.00001207 +/- 0.00000349 72.89 +/- 23.73 0.00000681 +/- 0.00000206
DATA: success (warning: decay rates inconsistent) 0.025 Belarusian Anatolia_Neolithic Uygur 3.85 3.45 2.16 37% 70.37 +/- 13.51 0.00001582 +/- 0.00000411 81.39 +/- 21.48 0.00001207 +/- 0.00000349 102.34 +/- 33.39 0.00001107 +/- 0.00000512
DATA: success (warning: decay rates inconsistent) 0.021 Belarusian British-Roman Koryak 3.89 2.37 2.28 74% 62.36 +/- 16.01 0.00003903 +/- 0.00000934 52.03 +/- 19.63 0.00002305 +/- 0.00000974 113.23 +/- 49.75 0.00002665 +/- 0.00001027
DATA: success (warning: decay rates inconsistent) 0.0084 Belarusian British-Roman Mixtec 4.11 2.37 2.50 80% 64.78 +/- 15.52 0.00004703 +/- 0.00001145 52.03 +/- 19.63 0.00002305 +/- 0.00000974 121.19 +/- 40.18 0.00002185 +/- 0.00000872
DATA: success (warning: decay rates inconsistent) 0.01 Belarusian Chukchi Mala 4.06 2.79 4.06 60% 172.83 +/- 30.55 0.00002691 +/- 0.00000663 110.36 +/- 39.54 0.00002861 +/- 0.00000981 93.18 +/- 21.71 0.00001222 +/- 0.00000301
DATA: success (warning: decay rates inconsistent) 0.047 Belarusian Itelmen Uzbek_WGA 3.69 2.36 2.20 54% 142.22 +/- 27.73 0.00006725 +/- 0.00001821 129.35 +/- 53.29 0.00003152 +/- 0.00001338 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.009 Belarusian LBK_EN Selkup 4.09 2.18 2.41 67% 67.83 +/- 16.58 0.00002655 +/- 0.00000641 115.11 +/- 38.65 0.00001960 +/- 0.00000899 57.06 +/- 20.02 0.00000933 +/- 0.00000386
DATA: success (warning: decay rates inconsistent) 0.05 Belarusian Selkup Turkish_Trabzon 3.68 2.41 2.11 32% 56.53 +/- 15.37 0.00001451 +/- 0.00000330 57.06 +/- 20.02 0.00000933 +/- 0.00000386 77.83 +/- 33.34 0.00000751 +/- 0.00000355
DATA: success (warning: decay rates inconsistent) 0.017 Belarusian Abhkasian Lahu 4.21 2.47 2.97 174% 32.04 +/- 6.68 0.00001002 +/- 0.00000238 3.95 +/- 1.60 0.00000098 +/- 0.00000024 57.34 +/- 19.33 0.00001384 +/- 0.00000369
DATA: success (warning: decay rates inconsistent) 0.00018 Belarusian Ami_Coriell Uzbek_WGA 5.15 2.09 2.20 63% 162.32 +/- 22.43 0.00007649 +/- 0.00001486 118.09 +/- 56.57 0.00002688 +/- 0.00001279 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.0041 Belarusian Anatolia_Neolithic Chukchi 4.52 3.45 2.79 35% 77.64 +/- 16.37 0.00003602 +/- 0.00000797 81.39 +/- 21.48 0.00001207 +/- 0.00000349 110.36 +/- 39.54 0.00002861 +/- 0.00000981
DATA: success (warning: decay rates inconsistent) 0.025 Belarusian Anatolia_Neolithic Daur 4.12 3.45 2.63 47% 73.39 +/- 17.81 0.00002378 +/- 0.00000569 81.39 +/- 21.48 0.00001207 +/- 0.00000349 118.84 +/- 40.98 0.00002486 +/- 0.00000947
DATA: success 0.05 Belarusian Anatolia_Neolithic Han 3.96 3.45 3.00 17% 79.39 +/- 18.74 0.00002687 +/- 0.00000678 81.39 +/- 21.48 0.00001207 +/- 0.00000349 93.68 +/- 31.25 0.00002137 +/- 0.00000623
DATA: success 0.00052 Belarusian Anatolia_Neolithic Han_NChina 4.94 3.45 3.58 22% 81.48 +/- 14.68 0.00003182 +/- 0.00000644 81.39 +/- 21.48 0.00001207 +/- 0.00000349 101.71 +/- 28.43 0.00002310 +/- 0.00000644
DATA: success 0.018 Belarusian Anatolia_Neolithic Miao 4.20 3.45 3.63 10% 78.02 +/- 16.13 0.00002457 +/- 0.00000585 81.39 +/- 21.48 0.00001207 +/- 0.00000349 86.31 +/- 23.79 0.00001726 +/- 0.00000411
DATA: success 0.0014 Belarusian Anatolia_Neolithic Turkmen 4.74 3.45 3.07 11% 76.01 +/- 10.03 0.00001185 +/- 0.00000250 81.39 +/- 21.48 0.00001207 +/- 0.00000349 72.89 +/- 23.73 0.00000681 +/- 0.00000206
DATA: success (warning: decay rates inconsistent) 0.00046 Belarusian Atayal_Coriell Uzbek_WGA 4.97 2.02 2.20 53% 179.16 +/- 31.95 0.00008213 +/- 0.00001654 130.82 +/- 54.40 0.00002576 +/- 0.00001275 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.027 Belarusian British-Roman Mixtec 4.11 2.37 2.50 80% 64.78 +/- 15.52 0.00004703 +/- 0.00001145 52.03 +/- 19.63 0.00002305 +/- 0.00000974 121.19 +/- 40.18 0.00002185 +/- 0.00000872
DATA: success (warning: decay rates inconsistent) 0.033 Belarusian Chukchi Mala 4.06 2.79 4.06 60% 172.83 +/- 30.55 0.00002691 +/- 0.00000663 110.36 +/- 39.54 0.00002861 +/- 0.00000981 93.18 +/- 21.71 0.00001222 +/- 0.00000301
DATA: success (warning: decay rates inconsistent) 0.009 Belarusian Dai Greek_Islands 4.35 3.05 2.28 32% 122.32 +/- 24.18 0.00004797 +/- 0.00001103 88.71 +/- 29.04 0.00001846 +/- 0.00000511 102.11 +/- 26.83 0.00001569 +/- 0.00000687
DATA: success (warning: decay rates inconsistent) 0.049 Belarusian Dai Uzbek_WGA 3.97 3.05 2.20 87% 160.47 +/- 30.16 0.00006276 +/- 0.00001582 88.71 +/- 29.04 0.00001846 +/- 0.00000511 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.0015 Belarusian Daur North_Ossetian 4.73 2.63 2.09 122% 42.94 +/- 8.92 0.00000724 +/- 0.00000153 118.84 +/- 40.98 0.00002486 +/- 0.00000947 178.58 +/- 51.90 0.00001887 +/- 0.00000901
DATA: success (warning: decay rates inconsistent) 0.047 Belarusian Daur Uzbek_WGA 3.97 2.63 2.20 62% 164.70 +/- 29.83 0.00008292 +/- 0.00002087 118.84 +/- 40.98 0.00002486 +/- 0.00000947 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.00086 Belarusian Eskimo_Chaplin LBK_EN 4.84 2.29 2.18 73% 53.65 +/- 11.08 0.00002657 +/- 0.00000479 63.81 +/- 27.89 0.00001618 +/- 0.00000586 115.11 +/- 38.65 0.00001960 +/- 0.00000899
DATA: success (warning: decay rates inconsistent) 0.027 Belarusian Georgian Lahu 4.10 2.89 2.97 166% 43.55 +/- 10.61 0.00001537 +/- 0.00000311 5.27 +/- 1.82 0.00000079 +/- 0.00000023 57.34 +/- 19.33 0.00001384 +/- 0.00000369
DATA: success (warning: decay rates inconsistent) 0.05 Belarusian Georgian Yi 3.96 2.89 3.26 179% 35.28 +/- 8.91 0.00000897 +/- 0.00000226 5.27 +/- 1.82 0.00000079 +/- 0.00000023 93.65 +/- 25.60 0.00002033 +/- 0.00000624
DATA: success 0.049 Belarusian Greek_Islands Han 3.96 2.28 3.00 15% 108.92 +/- 26.70 0.00004292 +/- 0.00001083 102.11 +/- 26.83 0.00001569 +/- 0.00000687 93.68 +/- 31.25 0.00002137 +/- 0.00000623
DATA: success 0.044 Belarusian Greek_Islands Miao 3.99 2.28 3.63 18% 102.98 +/- 23.71 0.00003805 +/- 0.00000953 102.11 +/- 26.83 0.00001569 +/- 0.00000687 86.31 +/- 23.79 0.00001726 +/- 0.00000411
DATA: success 0.0037 Belarusian Greek_Islands Mixtec 4.54 2.28 2.50 24% 95.62 +/- 12.99 0.00003944 +/- 0.00000868 102.11 +/- 26.83 0.00001569 +/- 0.00000687 121.19 +/- 40.18 0.00002185 +/- 0.00000872
DATA: success 0.045 Belarusian Greek_Islands Nganasan 3.98 2.28 2.06 5% 96.79 +/- 24.29 0.00003965 +/- 0.00000954 102.11 +/- 26.83 0.00001569 +/- 0.00000687 97.92 +/- 45.95 0.00001669 +/- 0.00000809
DATA: success 0.018 Belarusian Greek_Islands Ulchi 4.20 2.28 3.37 15% 118.40 +/- 24.18 0.00005248 +/- 0.00001249 102.11 +/- 26.83 0.00001569 +/- 0.00000687 114.38 +/- 33.90 0.00002845 +/- 0.00000805
DATA: success (warning: decay rates inconsistent) 0.042 Belarusian Greek_Islands Xibo 4.00 2.28 2.59 37% 101.05 +/- 22.65 0.00003689 +/- 0.00000922 102.11 +/- 26.83 0.00001569 +/- 0.00000687 70.25 +/- 27.10 0.00001649 +/- 0.00000507
DATA: success 0.0061 Belarusian Greek_Islands Yukagir_Tundra 4.44 2.28 2.55 18% 100.12 +/- 22.57 0.00004369 +/- 0.00000882 102.11 +/- 26.83 0.00001569 +/- 0.00000687 119.62 +/- 45.23 0.00002624 +/- 0.00001028
DATA: success (warning: decay rates inconsistent) 1.8e-05 Belarusian Han Uzbek_WGA 5.56 3.00 2.20 83% 145.83 +/- 21.23 0.00006518 +/- 0.00001171 93.68 +/- 31.25 0.00002137 +/- 0.00000623 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.023 Belarusian Han Yemenite_Jew 4.14 3.00 2.19 41% 101.08 +/- 19.78 0.00002665 +/- 0.00000644 93.68 +/- 31.25 0.00002137 +/- 0.00000623 142.22 +/- 61.15 0.00001902 +/- 0.00000870
DATA: success (warning: decay rates inconsistent) 0.00017 Belarusian Han_NChina Uzbek_WGA 5.15 3.58 2.20 76% 147.58 +/- 21.48 0.00006493 +/- 0.00001261 101.71 +/- 28.43 0.00002310 +/- 0.00000644 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.021 Belarusian Han_NChina Yemenite_Jew 4.16 3.58 2.19 47% 88.25 +/- 14.91 0.00002464 +/- 0.00000593 101.71 +/- 28.43 0.00002310 +/- 0.00000644 142.22 +/- 61.15 0.00001902 +/- 0.00000870
DATA: success (warning: decay rates inconsistent) 0.045 Belarusian Japanese Uzbek_WGA 3.99 2.51 2.20 76% 158.76 +/- 32.98 0.00007182 +/- 0.00001802 101.02 +/- 40.27 0.00002259 +/- 0.00000766 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.034 Belarusian Korean Uzbek_WGA 4.05 3.28 2.20 72% 147.50 +/- 22.94 0.00006552 +/- 0.00001618 106.54 +/- 29.46 0.00002451 +/- 0.00000748 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.0022 Belarusian Lahu Turkish_Jew 4.65 2.97 3.22 53% 53.69 +/- 9.97 0.00001763 +/- 0.00000379 57.34 +/- 19.33 0.00001384 +/- 0.00000369 92.56 +/- 21.52 0.00000780 +/- 0.00000242
DATA: success (warning: decay rates inconsistent) 7.8e-06 Belarusian Lahu Uzbek_WGA 5.70 2.97 2.20 119% 125.65 +/- 17.75 0.00006183 +/- 0.00001084 57.34 +/- 19.33 0.00001384 +/- 0.00000369 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.028 Belarusian Lahu Yemenite_Jew 4.10 2.97 2.19 85% 73.51 +/- 17.32 0.00002186 +/- 0.00000534 57.34 +/- 19.33 0.00001384 +/- 0.00000369 142.22 +/- 61.15 0.00001902 +/- 0.00000870
DATA: success (warning: decay rates inconsistent) 0.029 Belarusian LBK_EN Selkup 4.09 2.18 2.41 67% 67.83 +/- 16.58 0.00002655 +/- 0.00000641 115.11 +/- 38.65 0.00001960 +/- 0.00000899 57.06 +/- 20.02 0.00000933 +/- 0.00000386
DATA: success (warning: decay rates inconsistent) 3e-05 Belarusian Miao Uzbek_WGA 5.47 3.63 2.20 89% 141.79 +/- 17.01 0.00005964 +/- 0.00001090 86.31 +/- 23.79 0.00001726 +/- 0.00000411 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.012 Belarusian Miao Yemenite_Jew 4.29 3.63 2.19 49% 96.51 +/- 17.73 0.00002466 +/- 0.00000575 86.31 +/- 23.79 0.00001726 +/- 0.00000411 142.22 +/- 61.15 0.00001902 +/- 0.00000870
DATA: success (warning: decay rates inconsistent) 0.036 Belarusian Naxi Uzbek_WGA 4.04 2.35 2.20 87% 150.57 +/- 27.26 0.00006598 +/- 0.00001633 88.34 +/- 37.62 0.00001891 +/- 0.00000714 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.0037 Belarusian Oroqen Uzbek_WGA 4.54 2.50 2.20 75% 159.87 +/- 26.32 0.00007776 +/- 0.00001713 102.18 +/- 40.85 0.00002369 +/- 0.00000834 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.043 Belarusian She Uzbek_WGA 3.99 3.16 2.20 70% 177.32 +/- 34.01 0.00008208 +/- 0.00002055 108.68 +/- 31.62 0.00002238 +/- 0.00000708 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.029 Belarusian Tu Uzbek_WGA 4.09 2.28 2.20 85% 150.44 +/- 31.12 0.00006074 +/- 0.00001485 91.29 +/- 40.04 0.00001929 +/- 0.00000802 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.001 Belarusian Tujia Uzbek_WGA 4.80 2.09 2.20 61% 164.13 +/- 25.59 0.00008133 +/- 0.00001693 120.48 +/- 57.69 0.00002290 +/- 0.00001057 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success 0.022 Belarusian Tujia Yemenite_Jew 4.15 2.09 2.19 18% 118.59 +/- 25.68 0.00002955 +/- 0.00000711 120.48 +/- 57.69 0.00002290 +/- 0.00001057 142.22 +/- 61.15 0.00001902 +/- 0.00000870
DATA: success (warning: decay rates inconsistent) 0.0053 Belarusian Ulchi Uzbek_WGA 4.47 3.37 2.20 65% 153.49 +/- 25.35 0.00007000 +/- 0.00001567 114.38 +/- 33.90 0.00002845 +/- 0.00000805 225.56 +/- 61.89 0.00007507 +/- 0.00003406
DATA: success (warning: decay rates inconsistent) 0.00055 Belarusian Uzbek_WGA Xibo 4.93 2.20 2.59 105% 129.90 +/- 24.58 0.00005579 +/- 0.00001132 225.56 +/- 61.89 0.00007507 +/- 0.00003406 70.25 +/- 27.10 0.00001649 +/- 0.00000507
DATA: success (warning: decay rates inconsistent) 0.00062 Belarusian Uzbek_WGA Yi 4.91 2.20 3.26 83% 156.22 +/- 22.94 0.00007252 +/- 0.00001478 225.56 +/- 61.89 0.00007507 +/- 0.00003406 93.65 +/- 25.60 0.00002033 +/- 0.00000624
DATA: success (warning: decay rates inconsistent) 0.011 Belarusian Uzbek_WGA Yukagir_Tundra 4.31 2.20 2.55 61% 182.09 +/- 32.35 0.00008497 +/- 0.00001970 225.56 +/- 61.89 0.00007507 +/- 0.00003406 119.62 +/- 45.23 0.00002624 +/- 0.00001028
DATA: success (warning: decay rates inconsistent) 0.048 Belarusian Yemenite_Jew Yi 3.97 2.19 3.26 41% 130.87 +/- 22.50 0.00003478 +/- 0.00000876 142.22 +/- 61.15 0.00001902 +/- 0.00000870 93.65 +/- 25.60 0.00002033 +/- 0.00000624После отсеивания не очень пригодных для дальнейшего анализа триплетов у нас осталась следующие комбинации:
DATA: success 0.014 Belarusian Anatolia_Neolithic Mala 4.01 4.42 3.87 12% 81.9 +/- 17.24 0.00001547 +/- 0.00000385 92.46 +/- 19.49 0.00001427 +/- 0.00000323 87.55 +/- 18.75 0.00001071 +/- 0.00000277
DATA: success 0.00024 Belarusian Anatolia_Neolithic Turkmen 4.88 4.42 2.26 12% 81.66 +/- 10.18 0.00001288 +/- 0.00000264 92.46 +/- 19.49 0.00001427 +/- 0.00000323 85.64 +/- 28.96 0.00000855 +/- 0.00000378
DATA: success 0.00044 Belarusian Anatolia_Neolithic Turkmen 4.74 3.45 3.07 11% 76.01 +/- 10.03 0.00001185 +/- 0.0000025 81.39 +/- 21.48 0.00001207 +/- 0.00000349 72.89 +/- 23.73 0.00000681 +/- 0.00000206
DATA: success 0.05 Belarusian Anatolia_Neolithic Han 3.96 3.45 3 17% 79.39 +/- 18.74 0.00002687 +/- 0.00000678 81.39 +/- 21.48 0.00001207 +/- 0.00000349 93.68 +/- 31.25 0.00002137 +/- 0.00000623
DATA: success 0.00052 Belarusian Anatolia_Neolithic Han_NChina 4.94 3.45 3.58 22% 81.48 +/- 14.68 0.00003182 +/- 0.00000644 81.39 +/- 21.48 0.00001207 +/- 0.00000349 101.71 +/- 28.43 0.0000231 +/- 0.00000644
DATA: success 0.018 Belarusian Anatolia_Neolithic Miao 4.2 3.45 3.63 10% 78.02 +/- 16.13 0.00002457 +/- 0.00000585 81.39 +/- 21.48 0.00001207 +/- 0.00000349 86.31 +/- 23.79 0.00001726 +/- 0.00000411
DATA: success 0.0014 Belarusian Anatolia_Neolithic Turkmen 4.74 3.45 3.07 11% 76.01 +/- 10.03 0.00001185 +/- 0.0000025 81.39 +/- 21.48 0.00001207 +/- 0.00000349 72.89 +/- 23.73 0.00000681 +/- 0.00000206
DATA: success 0.049 Belarusian Greek_Islands Han 3.96 2.28 3 15% 108.92 +/- 26.7 0.00004292 +/- 0.00001083 102.11 +/- 26.83 0.00001569 +/- 0.00000687 93.68 +/- 31.25 0.00002137 +/- 0.00000623
DATA: success 0.044 Belarusian Greek_Islands Miao 3.99 2.28 3.63 18% 102.98 +/- 23.71 0.00003805 +/- 0.00000953 102.11 +/- 26.83 0.00001569 +/- 0.00000687 86.31 +/- 23.79 0.00001726 +/- 0.00000411
DATA: success 0.0037 Belarusian Greek_Islands Mixtec 4.54 2.28 2.5 24% 95.62 +/- 12.99 0.00003944 +/- 0.00000868 102.11 +/- 26.83 0.00001569 +/- 0.00000687 121.19 +/- 40.18 0.00002185 +/- 0.00000872
DATA: success 0.045 Belarusian Greek_Islands Nganasan 3.98 2.28 2.06 5% 96.79 +/- 24.29 0.00003965 +/- 0.00000954 102.11 +/- 26.83 0.00001569 +/- 0.00000687 97.92 +/- 45.95 0.00001669 +/- 0.00000809
DATA: success 0.018 Belarusian Greek_Islands Ulchi 4.2 2.28 3.37 15% 118.4 +/- 24.18 0.00005248 +/- 0.00001249 102.11 +/- 26.83 0.00001569 +/- 0.00000687 114.38 +/- 33.9 0.00002845 +/- 0.00000805
DATA: success 0.0061 Belarusian Greek_Islands Yukagir_Tundra 4.44 2.28 2.55 18% 100.12 +/- 22.57 0.00004369 +/- 0.00000882 102.11 +/- 26.83 0.00001569 +/- 0.00000687 119.62 +/- 45.23 0.00002624 +/- 0.00001028
DATA: success 0.022 Belarusian Tujia Yemenite_Jew 4.15 2.09 2.19 18% 118.59 +/- 25.68 0.00002955 +/- 0.00000711 120.48 +/- 57.69 0.0000229 +/- 0.00001057 142.22 +/- 61.15 0.00001902 +/- 0.0000087
На третьем этапе программа определяет статистическая значимость комбинации (p-статистику):
P-значение (англ. P-value) — величина, используемая при тестировании статистических гипотез. Фактически это вероятность ошибки при отклонении нулевой гипотезы (ошибки первого рода). Проверка гипотез с помощью P-значения является альтернативой классической процедуре проверки через критическое значение распределения.
Обычно P-значение равно вероятности того, что случайная величина с данным распределением (распределением тестовой статистики при нулевой гипотезе) примет значение, не меньшее, чем фактическое значение тестовой статистики.
Отберем значения P меньше 0.05
DATA: success 0.00024 Belarusian Anatolia_Neolithic Turkmen 4.88 4.42 2.26 12% 81.66 +/- 10.18 0.00001288 +/- 0.00000264 92.46 +/- 19.49 0.00001427 +/- 0.00000323 85.64 +/- 28.96 0.00000855 +/- 0.00000378
DATA: success 0.00044 Belarusian Anatolia_Neolithic Turkmen 4.74 3.45 3.07 11% 76.01 +/- 10.03 0.00001185 +/- 0.0000025 81.39 +/- 21.48 0.00001207 +/- 0.00000349 72.89 +/- 23.73 0.00000681 +/- 0.00000206
DATA: success 0.00052 Belarusian Anatolia_Neolithic Han_NChina 4.94 3.45 3.58 22% 81.48 +/- 14.68 0.00003182 +/- 0.00000644 81.39 +/- 21.48 0.00001207 +/- 0.00000349 101.71 +/- 28.43 0.0000231 +/- 0.00000644
DATA: success 0.0014 Belarusian Anatolia_Neolithic Turkmen 4.74 3.45 3.07 11% 76.01 +/- 10.03 0.00001185 +/- 0.0000025 81.39 +/- 21.48 0.00001207 +/- 0.00000349 72.89 +/- 23.73 0.00000681 +/- 0.00000206
DATA: success 0.0037 Belarusian Greek_Islands Mixtec 4.54 2.28 2.5 24% 95.62 +/- 12.99 0.00003944 +/- 0.00000868 102.11 +/- 26.83 0.00001569 +/- 0.00000687 121.19 +/- 40.18 0.00002185 +/- 0.00000872
DATA: success 0.0061 Belarusian Greek_Islands Yukagir_Tundra 4.44 2.28 2.55 18% 100.12 +/- 22.57 0.00004369 +/- 0.00000882 102.11 +/- 26.83 0.00001569 +/- 0.00000687 119.62 +/- 45.23 0.00002624 +/- 0.00001028
DATA: success 0.014 Belarusian Anatolia_Neolithic Mala 4.01 4.42 3.87 12% 81.9 +/- 17.24 0.00001547 +/- 0.00000385 92.46 +/- 19.49 0.00001427 +/- 0.00000323 87.55 +/- 18.75 0.00001071 +/- 0.00000277
DATA: success 0.018 Belarusian Anatolia_Neolithic Miao 4.2 3.45 3.63 10% 78.02 +/- 16.13 0.00002457 +/- 0.00000585 81.39 +/- 21.48 0.00001207 +/- 0.00000349 86.31 +/- 23.79 0.00001726 +/- 0.00000411
DATA: success 0.018 Belarusian Greek_Islands Ulchi 4.2 2.28 3.37 15% 118.4 +/- 24.18 0.00005248 +/- 0.00001249 102.11 +/- 26.83 0.00001569 +/- 0.00000687 114.38 +/- 33.9 0.00002845 +/- 0.00000805
DATA: success 0.022 Belarusian Tujia Yemenite_Jew 4.15 2.09 2.19 18% 118.59 +/- 25.68 0.00002955 +/- 0.00000711 120.48 +/- 57.69 0.0000229 +/- 0.00001057 142.22 +/- 61.15 0.00001902 +/- 0.0000087
DATA: success 0.044 Belarusian Greek_Islands Miao 3.99 2.28 3.63 18% 102.98 +/- 23.71 0.00003805 +/- 0.00000953 102.11 +/- 26.83 0.00001569 +/- 0.00000687 86.31 +/- 23.79 0.00001726 +/- 0.00000411
DATA: success 0.045 Belarusian Greek_Islands Nganasan 3.98 2.28 2.06 5% 96.79 +/- 24.29 0.00003965 +/- 0.00000954 102.11 +/- 26.83 0.00001569 +/- 0.00000687 97.92 +/- 45.95 0.00001669 +/- 0.00000809
DATA: success 0.049 Belarusian Greek_Islands Han 3.96 2.28 3 15% 108.92 +/- 26.7 0.00004292 +/- 0.00001083 102.11 +/- 26.83 0.00001569 +/- 0.00000687 93.68 +/- 31.25 0.00002137 +/- 0.00000623
Получаем следующие пары (с датировкой адмикса в поколениях и годах)
| Таргет | Референс 1 | Референс 2 | Поколения | Погрешность | Года | Погрешность | ||
| Belarusian | Anatolia_Neolithic | Turkmen | 85.64 | +/- | 28.96 | 2483.56 | +/- | 839.84 |
| Belarusian | Anatolia_Neolithic | Turkmen | 72.89 | +/- | 23.73 | 2113.81 | +/- | 688.17 |
| Belarusian | Anatolia_Neolithic | Han_NChina | 101.71 | +/- | 28.43 | 2949.59 | +/- | 824.47 |
| Belarusian | Anatolia_Neolithic | Turkmen | 72.89 | +/- | 23.73 | 2113.81 | +/- | 688.17 |
| Belarusian | Greek_Islands | Mixtec | 121.19 | +/- | 40.18 | 3514.51 | +/- | 1165.22 |
| Belarusian | Greek_Islands | Yukagir_Tundra | 119.62 | +/- | 45.23 | 3468.98 | +/- | 1311.67 |
| Belarusian | Anatolia_Neolithic | Mala | 87.55 | +/- | 18.75 | 2538.95 | +/- | 543.75 |
| Belarusian | Anatolia_Neolithic | Miao | 86.31 | +/- | 23.79 | 2502.99 | +/- | 689.91 |
| Belarusian | Greek_Islands | Ulchi | 114.38 | +/- | 33.9 | 3317.02 | +/- | 983.1 |
| Belarusian | Tujia | Yemenite_Jew | 142.22 | +/- | 61.15 | 4124.38 | +/- | 1773.35 |
| Belarusian | Greek_Islands | Miao | 86.31 | +/- | 23.79 | 2502.99 | +/- | 689.91 |
| Belarusian | Greek_Islands | Nganasan | 97.92 | +/- | 45.95 | 2839.68 | +/- | 1332.55 |
| Belarusian | Greek_Islands | Han | 93.68 | +/- | 31.25 | 2716.72 | +/- | 906.25 |
| Belarusian | Anatolia_Neolithic | Han | 93.68 | +/- | 31.25 | 2716.72 | +/- | 906.25 |
О чем свидетельствует результаты? Результаты указывают на наличие сигнала смешивания предковых популяций белорусов с неолитическими земледельцами (ближневосточные популяции и популяции ближнего Востока здесь выступают в качестве суррогата неолитических популяций), и с рядом восточноазиатских, сибирских и америндских популяций (здесь они выступают в качестве суррогата сибирского вклада в генофонд восточной Европы) cо средним интервалом смешения примерно 2850 +- 950 лет назад, т.е в период бронзового века.
Ниже приведены графики угасания LD в комбинации Anatolian-Neolithic + Mala
Затем я уменьшил масштаб подгонки (fitting) кривых угасания LD до 0.5 сантиморганид и взял в качестве референса палеогеномы с хорошим покрытием
Эксперимент с Loschbour + Stuttgart оказался неудачным.
Более правдоподобна комбинация андроновцев (Andronovo) и чукчей (дата адмикса — 125+-60 поколений тому назад)
Вторая попытка подгонки референсных популяций Loschbour и Stuttgart в качестве предковых групп оказался более удачным (дата адмикса — приблизительно 445 +- 56 поколений тому назад, времена энеолита)
Адмикс с сибирскими палеопопуляциями (MA1) примерно в два раза «моложе» (258 +- 42 поколения, бронзовый век)
Еще один вариант адмикса между палеолитическими жителями Европы и MA1 (датировка — 393 +- 75 поколений)
Теперь о поляках. К сожалению, результаты оказались гораздо более зашумленными, так как использовались данные генотипирования на платформе Illumina, имеющей меньшее пересечение снипов со снипами платформы Affymetrix HumanOrigins. Несмотря на это, программа ALDER нашла три комбинации, пусть и с несовместимой амплитудой угасания LD.
DATA: success (warning: decay rates inconsistent) 0.011 Pole Eskimo_Sireniki Irish-BA 3.87 2.01 2.53 40% 146.66 +/- 27.30 0.00035747 +/- 0.00009228 161.51 +/- 69.51 0.00013202 +/- 0.00006577 107.56 +/- 33.31 0.00015435 +/- 0.00006109
DATA: success (warning: decay rates inconsistent) 0.0068 Pole Eskimo_Sireniki Remedello_BA.SG 3.99 2.01 2.57 49% 110.88 +/- 21.02 0.00024049 +/- 0.00006022 161.51 +/- 69.51 0.00013202 +/- 0.00006577 182.60 +/- 39.99 0.00014922 +/- 0.00005796
DATA: success (warning: decay rates inconsistent) 0.035 Pole Eskimo_Chaplin Remedello_BA.SG 3.59 2.51 2.57 56% 102.38 +/- 21.46 0.00022199 +/- 0.00006181 126.26 +/- 42.96 0.00009643 +/- 0.00003846 182.60 +/- 39.99 0.00014922 +/- 0.00005796
Здесь тоже виден слабый сигнал адмикса популяций бронзового века из Западной Европы (Remedello и ирландского бронзового века) c америндскими популяциями.
Впрочем, дополнительный анализ в программе ROLLOFF (с уменьшенным масштабом подгонки — fitting) выдал правдоподобные (c низким значением P) варианты. Например, вариант Bichon + Georgian_Kakheti: 151.41 +/-38.18, p= 4.7e-06
Очень хорошим вариантом оказался вариант адмикса Esperstedt_MN-Halberstadt_LBA: (дата адмикса — 163.80 +/- 34.11), p=4.8e-07

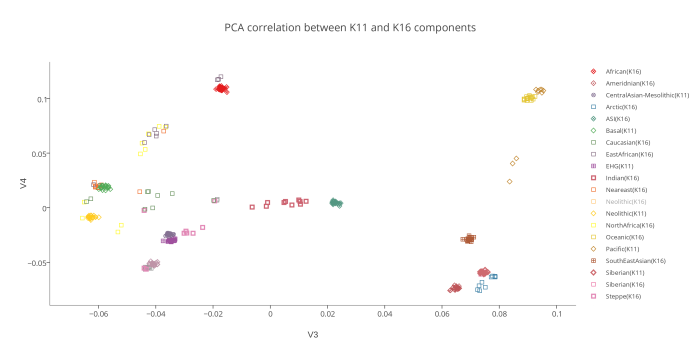 Вот эта таблица с усредненными значениями «симулянтов» компонентов K16 в калькуляторе K11 (колонки — компоненты K16, столбцы — компоненты K11, их пересечения — проекция компонентов K16 в компоненты K11).
Вот эта таблица с усредненными значениями «симулянтов» компонентов K16 в калькуляторе K11 (колонки — компоненты K16, столбцы — компоненты K11, их пересечения — проекция компонентов K16 в компоненты K11).


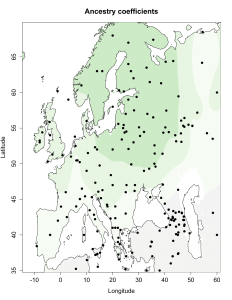
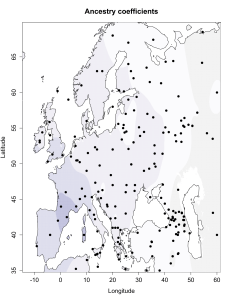
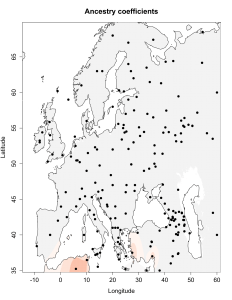
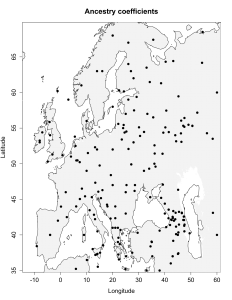








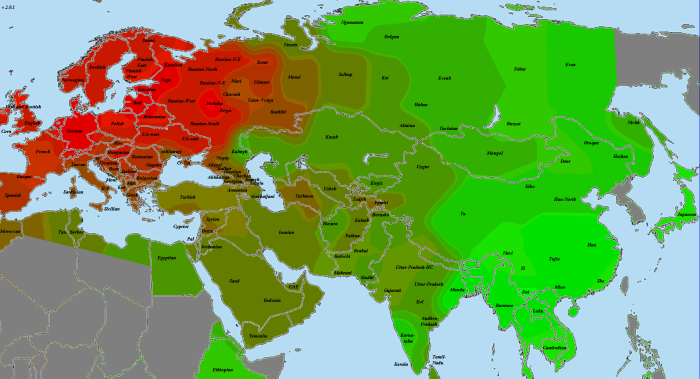
 При сравнении видно, что связь с восточной (и в первую очередь Юго-Восточной) Азией несколько ослабла, а связь с западными выборками (как европейскими, так и ближневосточными) слегка усилилась. Однако разница невелика и из-за этого сравнительная карта выглядит некрасиво. Чтобы избежать загромождения излишними иллюстрациями, ее не привожу.
При сравнении видно, что связь с восточной (и в первую очередь Юго-Восточной) Азией несколько ослабла, а связь с западными выборками (как европейскими, так и ближневосточными) слегка усилилась. Однако разница невелика и из-за этого сравнительная карта выглядит некрасиво. Чтобы избежать загромождения излишними иллюстрациями, ее не привожу.

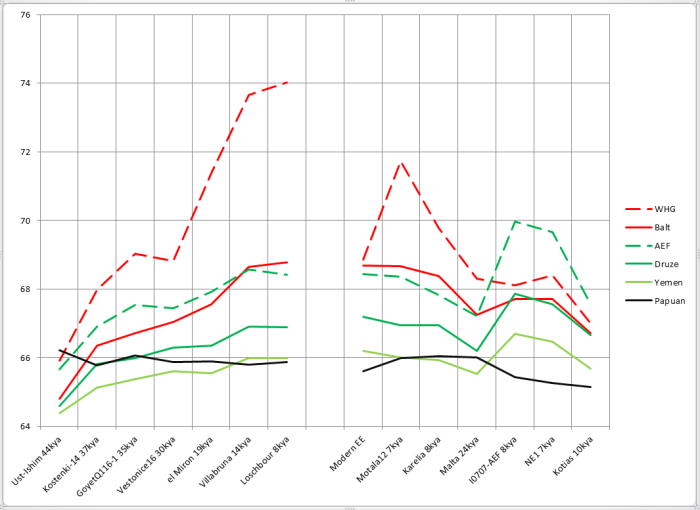
 Как видите, прибытие Villabruna никакого переворота не произвело. Как и раньше, с ходом времени сходство с циркумбалтийцами усиливалось, с восточноазиатами — ослабевало, с ближневосточниками — то ли слегка усиливалось, то ли медленно ослабевало, но медленнее, чем с восточноазиатами.
Как видите, прибытие Villabruna никакого переворота не произвело. Как и раньше, с ходом времени сходство с циркумбалтийцами усиливалось, с восточноазиатами — ослабевало, с ближневосточниками — то ли слегка усиливалось, то ли медленно ослабевало, но медленнее, чем с восточноазиатами.












Для отправки комментария необходимо войти на сайт.