Сергей Козлов
О «ближневосточном компоненте» палеолитических охотников-собирателей Европы
Описание
Рассмотрена статья Qiaomei Fu et al. «The genetic history of Ice Age Europe». Проведен анализ европейских палеогеномов возрастом от 37 до 8 тысяч лет из данной статьи и более ранних работ. Аутосомный компонент западных охотников-собирателей (WHG) — преимущественно результат генетического дрейфа, гипотеза авторов о его формировании в результате однократной миграции в Европу около 14 тысяч лет назад носителей ближневосточных аутосомных компонентов несостоятельна. Вместе с тем, обмен генофондом с ближневосточными популяциями несомненно происходил, однако для прояснения его истории необходимы палеогеномы с Ближнего Востока. Подтверждаются выводы из более старых работ о наличии ближневосточного («базального») компонента у образца Костенки-14 (человек с Маркиной Горы), отрицаемые в рассматриваемой статье. Вероятно, он связан с компонентом охотников-собирателей Кавказа (CHG). Опровергается вывод авторов о восточноазиатском влиянии на поздних WHG. Проведено моделирование ряда возможных событий смешения и построено дерево вероятных взаимосвязей аутосомных компонентов с размещением на нем имеющихся палеогеномов.
Обсуждение работы Qiaomei Fu et al на форуме «Молекулярная генеалогия».
Новые палеогеномы из статьи
В рассматриваемой статье впервые произведен временной срез геномов жителей Европы верхнего палеолита. Конечно, единичные геномы у нас были и раньше (Костенки-14, Oase1), однако не хватало системности для построения целостной картины изменений в генофонде европейцев на протяжении этого периода. Статья частично решает эту проблему — прочтено несколько десятков новых геномов. К сожалению, остался неохваченным период 19-28 тысяч лет назад (а с учетом лишь геномов приемлемого качества — 19-30 тлн), но и имеющиеся образцы позволяют сделать ряд интересных выводов.
Коротко о содержании рассматриваемой работы, критика
Авторы подтверждают выводы из более ранних работ об угасании вклада неандертальцев в генофонд современных европейцев с ходом времени (предположительно, на неандертальские участки ДНК действовал отрицательный отбор). Далее они касаются нескольких интересных мелочей (присутствие Y-гаплогруппы R1b в палеолитической Европе — образец Villabruna возрастом 14 тысяч лет, появление «мутации светлоглазости» почти одновременно в Европе и на Кавказе (разумеется, это не отменяет вероятности нахождения более древних образцов с этой мутацией впоследствии) и необычные для региона в наши дни митогаплогруппы). После этого авторы переходят к объединению образцов в кластеры и попытке реконструкции их взаимоотношений. По сути, здесь все просто — европейские палеогеномы из одной эпохи объединяются в один кластер. Классические европейские WHG выступают под псевдонимом «кластер Villabruna», их непосредственные предшественники — el Miron, и ряд геномов возрастом 30 тысяч лет (из них лишь один заслуживающего рассмотрения качества) — кластер Vestonice. Чуть более старые GoyetQ116-1 и костенковец не вошли ни в один кластер. Далее делается очень странный вывод, что с появлением кластера Villabruna (в дальнейшем я буду называть их «WHG» согласно общепринятой терминологии), произошло резкое изменение генофонда в результате вливания компонента, связанного с современными ближневосточными популяциями. Формально приводится и альтернативное объяснение — результат нормальной изменчивости среди охотников-собирателей, и группы с меньшей общностью с Ближним Востоком были замещены группами, изначально имевшими большую общность. Однако в abstract статьи попал лишь первый вариант.
Мое объяснение
Даже из диаграммы, которая должна иллюстрировать точку зрения авторов, следует прямо противоположный ей вывод — изменения, относимые к появлению классических WHG, начались задолго до этого и происходили постепенно. «Ближневосточное влияние» (зеленые ромбики) появляется в заметных масштабах уже в предшествующем кластере el Miron, на пять тысячелетий ранее. Но перед этим кластером находится разрыв в девять тысячелетий, где, вполне возможно, мы тоже могли бы увидеть это влияние. Однако на картинке разрыв закрыт и создается впечатление резкого перехода.
Исходное изображение:

Отмасштабированная пропорционально реальной временной шкале картинка:

Как я покажу в дальнейшем, общность палеообразцов с классическими WHG и современными северными европейцами (которые являются преимущественно потомками WHG) с ходом времени росла постоянно — от костенковца и GoyetQ116-1 к el Miron, Villabruna и Loschbour. По моим предположениям, основной механизм здесь — дрейф генов. Не надо думать, что это был некий целенаправленный процесс — наоборот, дрейф генов во многом случаен (хотя и отбор наверняка сыграл свою роль), но именно то, что получилось в его результате, и стало европейскими охотниками-собирателями мезолита. Поэтому естественно, что чем ближе к нашему времени, тем выше сходство с итоговым результатом процесса.
Вместе с тем, с ходом времени мы наблюдаем и относительное повышение общности с ближневосточными популяциями, хотя и в заметно меньшем масштабе. Однако трудно сказать, кто, когда, сколько раз и на кого влиял. Допустим в качестве модели, что несущие компонент WHG группы повлияли на ближневосточников в относительно недавнем прошлом. Тогда повышение сходства палеогеномов с WHG автоматически будет немного повышать сходство и с ближневосточниками пропорционально доле WHG в их генофонде, даже если в ту эпоху на Ближнем Востоке о WHG и не слыхали. С другой стороны, небольшие равномерные вливания с Ближнего Востока в Европу могли дать такой же эффект. Или же третья группа, вроде CHG, могла повлиять как на WHG, так и на ближневосточников (необязательно одновременно). Словом, точку здесь поставит лишь хорошая выборка палеогеномов с Ближнего Востока -сравнение с современными популяциями всегда оставит место гаданиям.
Что касается восточноазиатского влияния на часть WHG (внимательные читатели критикуемой работы могли заметить, что оно «проявляется» и у одного из древнейших образцов — GoyetQ116-1), то оно объясняется ошибочностью принятия основой для сравнения образца Kostenki-14. Далее я еще коснусь этого.
Использованные для анализа методы и палеообразцы, причины их выбора
В этой заметке я не стал применять свой излюбленный метод — подсчет сумм общих (IBD) сегментов. Хотя качество некоторых образцов вполне позволяет его применить, трудно понять, как при этом надежно сравнить между собой образцы из эпох, разделенных десятками тысячелетий? Ведь сегменты со временем уменьшаются в размерах, при этом скорость процесса сильно зависит от популяционной истории — в одной выборке быстрее, в другой медленнее… Добавим к этому резко различающееся качество прочтения палеогеномов, и за корректность сравнения поручиться становится совершенно невозможно.
Поэтому я решил пойти путем подсчета доли общих снипов (IBS), как простого и объективного показателя. Чем больше значений снипов совпадает, тем выше генетическая близость. Я не согласен с мнением, что учитывать надо лишь производные (derived) аллели — ведь если оба варианта закрепились в популяции, то для дрейфа генов уже безразлично, какой из них предковый. Для того, чтобы поставить геномы разного качества в одинаковые условия, я случайным образом выбрал для каждого аллеля одно из прочтений и оставил лишь его, то есть создал искусственную гаплоидность, как часто делается с палеогеномами от лаборатории Райха. Обычно я ругаю этот подход, как разрушающий IBD-сегменты, но в данном случае он приносит пользу. Далее я ограничил набор снипов пересечением трех множеств — снипы, используемые мной для сравнения с современными выборками и снипы, прочитанные у образцов Villabruna и GoyetQ116-1. Более логично было бы выбрать в качестве базового образца WHG прочитанный наиболее качественно из всех Loschbour, однако носитель R1b Villabruna в любом случае будет вызывать интерес общественности и подозрения в отличиях от других WHG, поэтому решение было принято в его пользу. Что касается GoyetQ116-1, то из всех древних образцов он наиболее связан с «промежуточным» между палеолитическими европейцами и WHG el-Miron, за что и был выбран в качестве второй опоры. Итоговый набор составил около 107 тысяч снипов. Для сравнения Villabruna и Goyet с el Miron было проведено отдельное уменьшение набора до присутсвующих у всех троих 65 тысячи снипов.
Среди остальных использованных палеообразцов хорошо прочитанные Loschbour, Ust-Ishim, Kostenki, NE1, Kotias отмасштабировались практически без потерь в количестве снипов, Mota1 и Motala12 — с незначительными потерями. Несколько хуже отмасштабировались Vestonice16, «карел» c Оленьего острова I0061, «мальтинец» и один из наиболее ранних геномов неолитчических земледельцев Анатолии I0707, но они также были включены в сравнение, поскольку представляют явный интерес. Судя по сравнению результатов I0707 и его близкого аналога из Европы NE1, подсчеты сохранили корректность.
Таблица результатов и ее применение
Результаты сравнения сведены в таблицу, с которой желающие могут ознакомиться по ссылке. Кроме современных выборок, приведены и выборки из имеющихся палеогеномов (конец таблицы), хотя их качество очень разное. Впрочем, интересующие нас в первую очередь западные охотники-собиратели WHG и ранние неолитические земледельцы Анатолии AEF представлены вполне неплохо, хотя по Анатолии пока, к сожалению, охвачена лишь крайняя западная часть. Наиболее древние европейцы — Kostenki14, GoyetQ116-1, Vestonice16 объединены в выборку pre-WHG. Число в каждой ячейке — доля совпадающих аллелей для текущего образца с этой выборкой — допустим, 65 означает 65% общих снипов (на данном наборе снипов — число сильно зависит от набора).
Несмотря на все ухищрения, призванные поставить геномы в равные условия, прямое сравнение результатов оказалось невозможным — у некоторых образцов чуть больше совпадающих снипов со всеми выборками, у некоторых — чуть меньше. Разница невелика, но в этом методе играют роль даже доли процента. Возможно, причина — в разном качестве прочтения, возможно — индивидуальные особенности образцов или что-то еще. Однако решение проблемы существует. Поскольку увеличение или уменьшение доли совпадающих снипов примерно пропорционально для всех выборок, можно взять соотношение этой доли с выборкой, равно удаленной от всех («outgroup»). В качестве подобного ориентира я решил взять объединение всех четырех используемых мной выборок из Африки южнее Сахары — представителей пигмеев мбути и бьяка, кенийских банту, нигерийского племени йоруба. На графике ниже приведена доля общих снипов для каждого из палеогеномов с соответствующей выборкой (Balt, Druze, WHG и т.д.) после приведения доли общих снипов с африканцами к одинаковому с другими образцами значению путем домножения на коэффициент. Для проверки корректности метода на график помещены другие outgroups, которые в исследуемый период явно не могли участвовать в обмене генами ни с африканцами, ни с исследуемыми палеообразцами — выборка папуасов. Как интерпретировать их результат, я опишу чуть ниже.
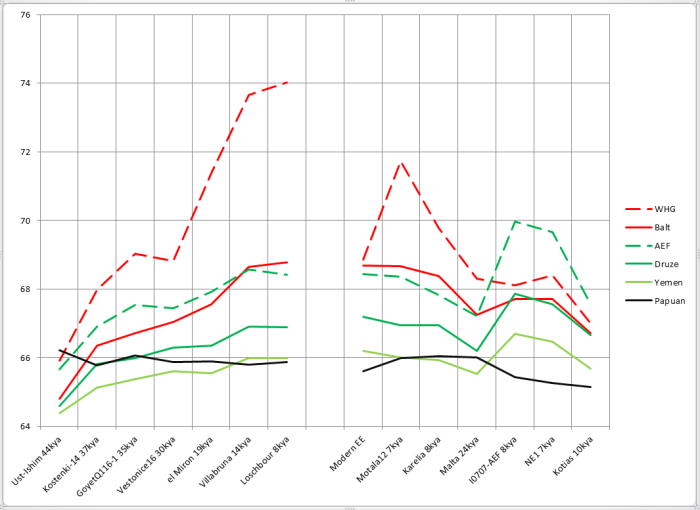
Палеогеномы (kya означает тысяч лет назад):
Ust-Ishim — усть-ишимский человек, наиболее древний приемлемо прочитанный геном человека современного типа.
Kostenki-14, GoyetQ116-1, Vestonice16 — древние геномы из Европы
el-Miron — предшественники WHG
Villabruna, Loschbour — WHG
Motala12 — охотник-собиратель из Швеции, представитель группы SHG (охотники-собиратели Скандинавии)
Karelian — образец с Оленьего Острова, так называемый EHG (восточный охотник-собиратель). Malta — древний «сибиряк» со стоянки Мальта, образец аутосомного компонента ANE — предковые северные евразийцы
EHG находятся в промежутке между WHG и ANE и, вероятно, являются их смесью.
I0707 — ранний неолитический земледелец с запада Анатолии
NE1 — ранний неолитический земледелец с территории Венгрии
Kotias — мезолитический охотник-собиратель с Кавказа
Ради интереса я также поместил на график результаты современного восточноевропейца с предками из трех восточнославянских народов (Modern EE).
Левая часть графика иллюстрирует изменения в генофонде европейцев с течением времени (усть-ишимский человек добавлен для сравнения, хотя он и не из Европы), правая — другие представляющие интерес геномы.
При сравнениях палеогеномов с палеовыборками сравнение «сам с собой» пропускалось.
Интерпретация сравнения с выборкой папуасов
Как мы видим, соотношение «родство с папуасами»/»родство с африканцами» для палеоевропейцев представляет собой почти горизонтальную линию. Это значит, что с какой скоростью европейцы «отдрейфовывали» от папуасов, примерно с такой же они отдалялись и от суб-сахарцев. Выглядит логично. Усть-ишимец выше всех, и это тоже логично — ведь он находится наиболее близко во времени к моменту расхождения папусов, восточноазиатов и WHG/ANE — значит, он и должен иметь относительно больше общего с папуасами. С другой стороны, для образца Kotias, имеющего много «базального» компонента, логично иметь заметно более низкое значение этого соотношения — момент расхождения «базальников» и предков остальных не-африканцев (включая папуасов) был очень давно. Ранние земледельцы, как смесь «базальников» и WHG, закономерно находятся в промежутке между WHG и Kotias. Даже неравномерности в графике охотников-собирателей находят свое объяснение — как я покажу позже, у костенковца вероятно небольшое влияние «базальников», и он проваливается на графике. Также я предполагаю небольшое базальное влияние у WHG и el Miron — соответственно, они находятся чуть ниже Goyet, мальтинца и оленеостровца. Итак, контрольная проверка показала применимость метода.
Важная ремарка — когда я в дальнейшем буду писать о росте доли общих снипов (график с течением времени идет вверх), надо понимать, что этот рост относительный. Есть некий базовый «уровень разбегания» — это скорость, с которой мы с каждым поколением отдаляемся от африканцев и папуасов из-за дрейфа генов и других факторов. Если в относительных значениях общность с друзами растет, это не значит, что она точно растет в абсолютных значениях — возможно, она тоже падает, но из-за обмена генами с нами падает медленнее, чем могла бы. А может, с друзами общность медленно растет, но с отстающими от них йеменцами медленно падает. Все зависит от соотношения скорости дрейфа генов, который нас растаскивает, и скорости обмена генами, который объединяет. В данном случае нас интересует, что удается увидеть наличие факта этого обмена.
Интерпретация графика
В первую очередь бросается в глаза пунктирная красная линия вверху — доля общих снипов с выборкой WHG. Как легко заметить, рост был почти непрерывен в течение всего времени, лишь, немного споткнувшись на образце Vestonice (возможно, поэтому в статье отнесли этот кластер к «тупиковой ветви». Впрочем, на сравнении с балтской выборкой такого не происходит, а современные выборки все же качеством на порядок выше — значит, доверия им больше). Ниже сплошной красной линией приведено сравнение с наиболее близкой к WHG выборкой наших современников — жителями восточного побережья Балтики (выборка Balt состоит из 11 литовских образцов, 6 латышских, 2 из Латгалии и одного с российско-латышской границы). Здесь картина аналогична — каждый следующий во времени образец ближе к балтам, чем предыдущий, включая даже Vestonice16. Очевидно, что объяснить это монотонное приближение единоразовой миграцией невозможно, а вот процессы генетического дрейфа укладываются в модель замечательно. Зеленые линии — аналогичная пара для неолитических земледельцев (пунктир) и считающихся (по результатам аутосомного анализа) наряду с армянами их наиболее сохранившимися представителями на Ближнем Востоке друзами Палестины. Здесь мы тоже видим рост, но более медленный по сравнению с ростом сходства с WHG. Если учесть, что порядка четверти генофонда AEF считается полученным от WHG, то примерно половину роста необходимо отнести на этот фактор. Оставшаяся половина и будет искомым обменом генами между «базальниками» и WHG. Для моделирования «базальников» зачастую применяют выборку из Йемена, как наиболее отдаленную от европейцев среди ближневосточников. Неизвестно, насколько это моделирование корректно, однако я включил их в сравнение (голубая линия). Родство с ними также растет, хотя и медленнее, чем с AEF или друзами. Однако, начав заметно ниже папуасов, ближе к нашему времени йеменцы успешно обгоняют их и становятся более близкими к WHG. Ведь обмен генами с йеменцами гораздо менее затруднен географически, чем с папуасами.
Несколько слов о правой половине графика
Представитель сестринской к WHG клады — ANE, мальтинец (24 тлн), обладает относительным сродством с WHG примерно на уровне европейских образцов 30-37 тысяч лет назад. Можно предположить, что момент расхождения был не слишком задолго до этого времени. При этом сродство с «балтской» выборкой относительно выше — поскольку в Восточной Европе присутствует не только WHG, но и доля ANE. У «карела» EHG связь с WHG закономерно выше (поскольку он и сам частично WHG), соответственно выросла и связь с ближневосточниками. То же самое, но в еще большей степени можно сказать про образец из Швеции Motala12 (скандинавские охотники-собиратели — SHG считаются WHG с примесью ANE). На паре AEF/NE1 можно пронаблюдать, как при продвижении в Европу у неолитчиков вырос вклад WHG, зато упал «ближневосточный» компонент. У «палеокавказца» Kotias по сравнению с ними резко падает связь с восточноевропейцами, и менее резко, но тоже падает — с ближневосточниками.
Определенный интерес представляет и сравнение с некоторыми другими современными выборками. Я не стал помещать их на основной график, чтобы избежать его перегруженности, но размещаю более полный вариант ниже.

Сардинцы добавлены, как наиболее яркие современные представители неолитических земледельцев, удмурты — как связанные с EHG, корнцы — с более западным вариантом WHG, калаши — за «калашский» кластер, кеты и южноамериканские индейцы каритиана — за связь с ANE.
Карты для палеогеномов
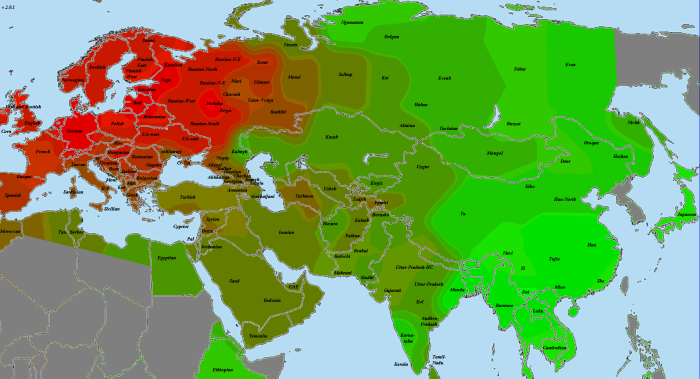
Теперь перейдем к рассмотрению каждого из палеогеномов отдельно. Для начала несколько слов об усть-ишимце. Хотя он и наиболее близок к общему корню, но все же, судя по всему, в его времена расхождение неафриканского человечества на основные ветви уже состоялось. Ближайшими к усть-ишимцу выборками оказались меланезийцы и папуасы, далее идут жители юго-восточной Азии, тамилы и восточноазиаты.
Каждая карта нормируется отдельно — ярко-красным выделяется наиболее хорошо связанная с этим геномом выборка из представленных, ярко-зеленым — наименее связанная. Не представленные на карте выборки (четыре африканские, две америндские, папуасы и меланезийцы) в нормировании не участвуют, по сравнению с африканцами все неафриканцы были бы просто разными оттенками красного. Карты в этой статье построены согласно доле общих снипов (IBS), по тем же таблицам, что и предыдущий график. Это не IBD-анализ. В более хорошем качестве карты можно загрузить отсюда

Хотя европейцы и среднеазиаты чуть ближе к усть-ишимцу, чем североафриканцы и ближневосточники, разница сравнительно невелика. Частично удаление европейцев от усть-ишимца следует отнести на влияние «базальников», но думаю, WHG и сами по себе успели хорошо удалиться от восточной ветви человечества. Поэтому на роль представителя общей для всех базы усть-ишимец не годится.
GoyetQ116-1
По причинам, описанным мной в разделе «Использованные для анализа методы и палеообразцы», из наиболее древних европейских геномов на роль «базового» был выбран GoyetQ116-1. И, как показывает карта, уже 35 тысячелетий назад европейские аутосомы начали приобретать свои основные черты. На первом месте по схожести — уже упоминавшаяся выборка «Balt», она будет попадаться нам вновь и вновь. Родство с остальными европейцами выражено вполне отчетливо. Однако интересно обратить внимание на другие регионы. Во-первых, родство с североафриканскими и ближневосточными популяциями находится на том же уровне, что и родство с восточноазиатами. Видимо, мы поймали тот момент, когда протоевропейцы были равноудалены от этих двух стволов. В дальнейшем родство с восточноазиатами будет ослабевать, а с ближневосточниками — усиливаться. Как говорится, «география-это судьба».

Еще раз повторюсь, что речь идет о современных ближневосточниках. Насколько они репрезентативны по сравнению с населением региона 10, 20, 50 тысяч лет назад — совершенно непонятно.
Очень интересно «вторичное пятно» в Индии. Вероятно, оно было бы соединено яркой полосой с европейским ареалом, если бы не размывшие ее миграции «базальников» с юго-запада и восточноазиатов с северо-востока. При этом в юго-восточной Индии и Бирме ареал связи с прото-WHG перекрывается с ареалом хорошей связанности с усть-ишимцев. Не отсюда ли когда-то разошлись две наших ветки? Я не являюсь специалистом по Y-гаплогруппам, но кажется, с максимумом разнообразия макрогаплогруппы K, включающей в себя в качестве ветвей такие известные гаплогруппы, как N, O, R, Q, это соотносится хорошо (в таком случае, «базальников» можно связать с IJ). Разумеется, сюда также относится оговорка о возможной несхожести современного и древнего населения.
Vestonice16
Картина для Vestonice16 довольно схожа с картой GoyetQ116-1.
 При сравнении видно, что связь с восточной (и в первую очередь Юго-Восточной) Азией несколько ослабла, а связь с западными выборками (как европейскими, так и ближневосточными) слегка усилилась. Однако разница невелика и из-за этого сравнительная карта выглядит некрасиво. Чтобы избежать загромождения излишними иллюстрациями, ее не привожу.
При сравнении видно, что связь с восточной (и в первую очередь Юго-Восточной) Азией несколько ослабла, а связь с западными выборками (как европейскими, так и ближневосточными) слегка усилилась. Однако разница невелика и из-за этого сравнительная карта выглядит некрасиво. Чтобы избежать загромождения излишними иллюстрациями, ее не привожу.
Kostenki14
Как и Вестонице, костенковец весьма схож с GoyetQ116-1. В данном случае мне хочется привести именно карту разницы со вторым палеогеномом, чтобы продемонстрировать его «южный» компонент. Зеленое — больше общего с костенковцем, красное — с Goyet.

Из-за схожести двух геномов карта очень зашумлена, однако противоположности проявляются хорошо. Ярко-зеленое прекрасно совпадает с областью распространения компонента кавказских охотников-собирателей CHG (ниже будет приведена карта и для них). Видны его максимумы на Кавказе и у калашей, на Балканах, и даже (хотя это может быть погрешностью) замечавшееся при анализе «ямных» геномов пятно в северо-западной Европе. Красное же в юго-восточной Азии — район максимальной «небазальности». Оттенки бурого и близкие к ним разглядывать нет смысла, также, как и отдельные «выбросы».
Как будет показано далее, костенковец наиболее успешно моделируется, как смесь 86% GoyetQ116-1 и 14% Kotias. Строго говоря, мы не можем утверждать, что GoyetQ116-1 представляет чистых прото-WHG, а костенковец является смесью с южанами. Не исключено, что «южный» компонент присутствует и у GoyetQ116-1, просто его меньше. В конце концов, смешение могло произойти еще по пути в Европу.
el Miron
Закончив с наиболее древними геномами, мы можем перейти к рассмотрению динамики европейского генофонда во времени (впрочем, до момента прибытия неолитических земледельцев она довольно однообразна). Поэтому ближайшие карты будут только сравнительными. Итак, красное — выборки, сходство с которыми у образца el Miron (19 тлн) усилилось по сравнению с образцом GoyetQ116-1 (35 тлн). зеленое — выборки, сходство с которыми ослабло. Бурое — возможно, слегка усилилось, возможно, ослабло, но не так сильно, как с зеленым. Об этом я написал в разделе «важная ремарка» после графика.

Villabruna
 Как видите, прибытие Villabruna никакого переворота не произвело. Как и раньше, с ходом времени сходство с циркумбалтийцами усиливалось, с восточноазиатами — ослабевало, с ближневосточниками — то ли слегка усиливалось, то ли медленно ослабевало, но медленнее, чем с восточноазиатами.
Как видите, прибытие Villabruna никакого переворота не произвело. Как и раньше, с ходом времени сходство с циркумбалтийцами усиливалось, с восточноазиатами — ослабевало, с ближневосточниками — то ли слегка усиливалось, то ли медленно ослабевало, но медленнее, чем с восточноазиатами.
Loschbour
Этот образец настолько схож с предыдущим (см график), что разностная карта показывает один шум. Поэтому я приведу конечный итог — вот к чему пришли WHG спустя 29 тысячелетий:

А также сравнение — где произошли наибольшие изменения
Сравнение Loschbour и GoyetQ116-1

Дальше всего «убежали» от протоевропейцев жители юго-восточной Азии, далее идут Индия, Восточная Сибирь и Северная Африка. За пределами основного региона меньше всего «скорость убегания» на Северном Кавказе, у ираноязычных памирцев, греков-киприотов и кетов (везде можно предположить контакты с носителями WHG).
Теперь перейдем к Кавказу и Анатолии. Уже упоминавшийся в пояснениях к карте для костенковца кавказский охотник-собиратель Kotias:

Интересно попытаться расщепить этот компонент на составляющие. В значительной части он несомненно связан общим корнем с прото-WHG (хорошо выделяются оба значимых для этого компонента региона — Европа и Индия). Попробуем вычленить не-WHG часть путем сравнения с GoyetQ116-1.

В первую очередь закономерно выделяются зоны наибольшего распространения CHG — Кавказ и Афганистан (калаши)/Пакистан/Иран. Однако кроме этого, проявляется и связь с Ближним Востоком, Анатолией, Балканами — регионами распространения ранненеолитических земледельцев. Таким образом, можно предположить, что у CHG имеется связь с ближневосточным аутосомным компонентом (знаменитые «базальники»), который впоследствии стал основой генофонда неолитических земледельцев и через них повлиял на современных европейцев. Потому-то Европа и выглядит на этой карте в целом нейтрально — на юго-востоке персиливает влияние «базальников», на северо-востоке — WHG. И наоборот, Восточная Азия, куда базальники не добрались, оказалась ярко-зеленой — это говорит о том, что время их расхождения с восточноазиатами древнее, чем время расхождения восточноазиатов и WHG.
Тот же самый эффект, но с противоположной стороны мы можем наблюдать, сравнив Kotias и геном ранненеолитического земледельца из Анатолии:
Поскольку теперь Kotias менее «базальный», на этот раз Восточная Азия оказалась красной. Хотя наиболее выражен «не-базальный» компонент Kotias в Индии. Поэтому я считаю, что компонент CHG следует считать смешанным между «ближневосточным» (предковым к AEF) и «индийским» (предковым к WHG) компонентом.
Раз уж я неоднократно упомянул AEF, приведу карту и для представителя этой выборки I0707.

Среди наших современников наиболее схожими с ним являются жители острова Сардиния, находящемся в западной части Средиземного Моря. Можно сказать, что компонент ранних земледельцев сохранился там, словно в заповеднике. В целом он лучше представлен в южной Европе, чем на Ближнем Востоке. Хотя не стоит забывать — для анализа у нас есть лишь палеогеномы с крайнего запада Анатолии, на границе с Европой. Вполне возможно, что ближневосточные геномы оказались бы ближе к современным выборкам с Ближнего Востока. Пока же мы можем сказать, что в регионе наиболее схожими с имеющимися образцами неолитчиков оказались армяне, друзы и греки-киприоты.
Наконец, последними я хочу привести две карты для образца возрастом в 24 тысячелетия со стоянки Мальта в Прибайкалье. На основе его анализа в свое время было выдвинуто предположении о существовании «популяции-призрака» — ANE, предковых северных евразийцев, которые повлияли на многих соседей, в том числе на американских индейцев, но сами к нашему времени исчезли. ANE считаются родственной к WHG веткой и не несут восточноазиатского или ближневосточного влияния. В схожести картин можно легко убедиться:

Если WHG это западный вариант, то у ANE основная тяжесть приходится на выборки из Западной Сибири (кеты), Урала (манси) и недавных мигрантов из этого же региона (саами). Очевидно, в прошлом ареал ANE простирался заметно восточнее, но к нашим дням они оказались вытеснены мигрантами с юга, из Восточной Азии. Интересно сравнить, каковы же основные отличия ANE от прото-WHG:

Пятно в западной Сибири вполне ожидаемо. Меня более заинтересовало пятно вокруг выборки калашей в средней Азии. Если вспомнить о связи этого же региона с кавказскими охотниками-собирателями, то уместно предположить, что здесь мы нащупали корень не-ближневосточной части CHG. При анализе Admixture мальтинец показывал наличие около 30% CHG, поэтому я долго ломал голову, как связать этот факт с явной не-ближневосточностью мальтинца. Теперь все становится на свои места — взаимосвязь идет через «калашский» компонент.
Что касается отличий прото-WHG от ANE, то они чуть ближе к восточноазиатам (может, их точка отделения чуть юго-восточнее, чем у ANE?), и ближе к «базальникам», что вновь заставляет меня думать о «базальном» влиянии уже у GoyetQ116-1. В конце концов, если у двух других образцов оно есть, может быть и у этого. Но пока более «чистых» образцов у нас нет, сравнить не с кем. С другой стороны, мальтинский образец на одиннадцать тысячелетий моложе — возможно, за это время он сильнее отдрейфовал от остальных веток.
Численная оценка доли вклада каждого компонента в некоторые из адмиксов.
В процессе работы над сравнительными картами у меня возникла мысль, не попробовать ли сделать численную оценку на основе все тех же таблиц общности IBS с современными выборками. Действительно, если я предполагаю, что не-WHG компонент костенковца очень похож на результаты кавказского охотника-собирателя Kotias, то я могу проверить, насколько близка к костенковцу будет комбинация 1% Kotias + 99% GoyetQ116-1, 2% Kotias + 98% GoyetQ116-1 и так далее, проверив сумму среднеквадратичных отклонений по всем столбцам. Для того, чтобы исключить влияние уже упоминавшегося в начале статьи эффекта, для каждой тройки сравниваемых геномов производилось нормирование. Таким образом, суммы IBS с современными выборками по каждому геному совпадали.
Для проверки модели я решил использовать геном, смешанное происхождение которого достоверно известно. Как мы знаем, по мере продвижения в Европу и с ходом тысячелетий исходный генофонд неолитических земледельцев постепенно размывался благодаря влиянию местных охотников-собирателей. Следовательно, геном семитысячелетней давности земледельца из Венгрии NE1 должен хорошо моделироваться, как смесь земледельца из Анатолии AEF (возраст генома на тысячу лет больше) и WHG. Так и получается — если в роли представителя WHG выступает более ранний геном Villabruna, модель предсказывает соотношение 11% WHG на 89% AEF, для более позднего Loschbour соотношение почти такое же — 10% WHG на 90% AEF. Среднеквадратичное отклонение при этом меньше единицы — в дальнейшем будем считать такое значение признаком того, что смешение моделируется хорошо.
Ряд результатов для заинтересовавших меня вариантов моделирования приведен на изображениях ниже:

Кратко прокомментирую. При попытке смоделировать NE1, как смесь WHG и CHG отклонение резко возрастает, что говорит о неудачности такой модели по сравнению с предыдущим вариантом. Родственные WHG охотники-собиратели ANE могут частично служить заменой Villabruna, однако результат хуже. Таким образом, результаты моделирования полностью соответствуют здравому смыслу. Я решил попробовать сделать еще один шаг и ввести в модель искусственный образец «базальника», полученный вычитанием из геномов неолитических земледельцев 15-20 процентов вклада WHG. Конечно, точная доля компонента WHG в геномах неолитчиков нам неизвестна, однако это лучше, чем применять в качестве «базального» образца геном AEF.
Результат костенковца действительно лучше всего моделируется, как смесь 86% прото-WHG и 14% CHG (Kotias), что мы и наблюдали на сравнительной карте. Чуть хуже вариант 94% прото-WHG на 6% базальников. Для другого древнего образца из Европы, Vestonice16, картина противоположная — базальники лучше подходят в качестве второй стороны, чем кавказцы. Интересно, что наиболее старые образцы Y-гаплогруппы I пока что найдены именно у представителей кластера Вестонице — возможно, это не случайное совпадение и вливание «базального» компонента связано с приходом носителей этой гаплогруппы.
«Опорный» прото-WHG GoyetQ116-1 не моделируется, как смесь кого-либо из двух других представителей группы и южан. Однако он может быть относительно неплохо смоделирован, как 88% костенковца и 12% мальтинца. Вероятно, это связано с отсутствием «базального» компонента у образца со стоянки Мальта.

Носитель R1b Villabruna может быть смоделирован, как смесь одного из своих предшественников и базальников, однако отклонение при этом слишком велико, чтобы считать моделирование успешным.
CHG Kotias плохо моделируется, как смесь каких-либо двух других образцов. Наиболее удачный вариант — 48% Мальта и 52% базальники (что еще раз говорит о его промежуточном положении между двумя кладами).
«Оленеостровец» EHG наиболее хорошо моделируется, как смесь 51% SHG (Motala12) и 49% ANE (мальтинец), отклонение великовато.

«Скандинав» Motala12 хорошо моделируется, как смесь 72% WHG и 28% EHG
Промежуточный между прото- и классическими WHG образец el Miron оптимально моделируется именно как смесь первых (GoyetQ116-1) и вторых (Villabruna). Однако при этом он оказывается ближе к более древним родственникам, хотя расстояние по времени до них гораздо больше. Возможно, это объясняется ускорением дрейфа в эпоху 19-14 тлн, но мне кажется более правдоподобным другое объяснение — WHG это потомки сестринской к el Miron ветви, поэтому часть дрейфа у них прошла отдельно.
Дерево вероятных взаимосвязей
Попытавшись максимально подробно и непротиворечиво свести вместе как данные, полученные в результате вышеописанных исследований, так и информацию из других работ, я изобразил дерево возможных взаимодействий палеообразцов и аутосомных компонентов. Схема достаточно условна, поэтому размещать на ней датировки далее 40 тысяч лет назад не имеет смысла. Гипотетический общий компонент «мальтинца» и охотников собирателей-кавказа я обозначил «Kalash», но надо понимать, что под этим вовсе не подразумеваются современные калаши — просто неким образом связанная с ними древняя предковая популяция. Серыми стрелками между «базальниками» и CHG, «базальниками» и WHG обозначено, что взаимодействия, по-видимому, были, но обозначить их одиночной линией на схеме тяжело. «Уральский» компонент — это часть генофонда народов Урала и западной Сибири, которую можно отнести к европейской ветви, для получения картины современного состояния необходимо объединить ее с восточноазиатским влиянием.

Думаю, что на самом деле все гораздо сложнее и запутаннее, чем изображено здесь )) Будем ждать новых расшифровок древних геномов для дальнейшего развития схемы.











 При сравнении видно, что связь с восточной (и в первую очередь Юго-Восточной) Азией несколько ослабла, а связь с западными выборками (как европейскими, так и ближневосточными) слегка усилилась. Однако разница невелика и из-за этого сравнительная карта выглядит некрасиво. Чтобы избежать загромождения излишними иллюстрациями, ее не привожу.
При сравнении видно, что связь с восточной (и в первую очередь Юго-Восточной) Азией несколько ослабла, а связь с западными выборками (как европейскими, так и ближневосточными) слегка усилилась. Однако разница невелика и из-за этого сравнительная карта выглядит некрасиво. Чтобы избежать загромождения излишними иллюстрациями, ее не привожу.

 Как видите, прибытие Villabruna никакого переворота не произвело. Как и раньше, с ходом времени сходство с циркумбалтийцами усиливалось, с восточноазиатами — ослабевало, с ближневосточниками — то ли слегка усиливалось, то ли медленно ослабевало, но медленнее, чем с восточноазиатами.
Как видите, прибытие Villabruna никакого переворота не произвело. Как и раньше, с ходом времени сходство с циркумбалтийцами усиливалось, с восточноазиатами — ослабевало, с ближневосточниками — то ли слегка усиливалось, то ли медленно ослабевало, но медленнее, чем с восточноазиатами.












Для отправки комментария необходимо войти на сайт.