В этом посте мы продолжим обсуждение существующих методик и инструментов анализа т.н «локального происхождения» отдельных сегментов хромосом в человеческом геноме (под локальным происхождением здесь подразумевается предпологаемое географическое происхождение дискретного сегмента одной их двух парных аутосомных хромосом в геноме человека).
Ранее эта тема поднималась в описании программы SupportMix, а также в сжатом изложении методологии оценки происхождения хромосомных сегментов (инструмент PCAdmix). Данная заметка будет посвящена третьему инструменту — LAMP (Local Ancestry in adMixed Populations) (Sankararaman et al.2008).
Очевидно, что алгоритмы определения локального происхождения отдельных сегментов человеческих хромосом могут дать неплохие результаты при комбинированном использовании программ PLINK /ADMIXTUIRE/LAMP: например, комбинация этих программ позволяет довольно точно определить не только стратификацию отдельных этно-популяционных групп, но также и уровень «адмикса» у отдельных людей. Поскольку одна из задач нашего проекта MDLP состояла в определении практических и теоретических преимуществ и/или ограничений конкретных методологий биоинформатического анализа полных генома, я провел эксперимент, позволяющий прояснить ряд ограничений, которые значительно уменьшают уровень достоверности результатов субструктуры аутосомного генофонда населения Европы.
В качестве инструмента контроля качества комбинированного набора данных (аутосомных SNP-ов 22 хромосом) я использовал Plink, с помощью которого я выбрал для последующего анализа только качественные снипы (99% генотиприрования), частоты минорных аллелей которых превышают 1%.
Поскольку этно-популяционный фон неравновесного линикиджа марекеров (LD) может существенным образом влиять на основные компоненты субструктуры популяции, я исключил из выборки маркеры, характеризующиеся статистически значимым уровнем LD (с коэффициентом попарной корреляции r2 Пирсона > 0,4) в «скользящем окне» из 100 снипов с пошаговым сдвигом на 10 снипов. Кроме этого, я также использовал другие методы Plink для получения однородной выборки — например, кластеризации на основе IBS для обнаружения пары индивидов (outliers) с уровнем «родства», значительно более высоким, чем у пары выбранных случайным образом индивидов в однородной популяции. Под более высоким родством здесь понимается резко отклоняющиеся значения (более 3 стандартных отклонений) парных значнений IBS по отношению к остальной части выбаки, а также случаи с высоким значения PIHAT (более 0,05) и высокой степень инбридинга (гомозиготности*). Индивиды с подобными аномальными значениями («выбросы») были удалены из «обучающего» подмножества нашей выборки .
—
* В программе Plink степень инбридинга определяется через вероятностную функцию гомозиготности.
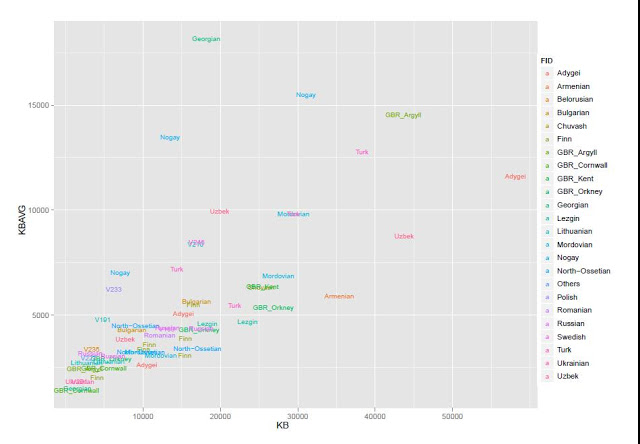

По окончанию описанных выше процедур фильтрации снипов и удаления «выбросов», окончательный набор данных представлял собой набор данных из 90 455 снипов и 317 человек (289 мужчин, 82 женщин). Эти данные были использованы в последующем анализе.
Прежде всего, мы использовали программу ADMIXTURE (Alexandre, Novembre, Lange 2009), в которой реализована модель оценки максимального правдоподобия (ML), т.е алгоритм кластеризации и оценки структуры популяции в наборе генетических данных (снипов).
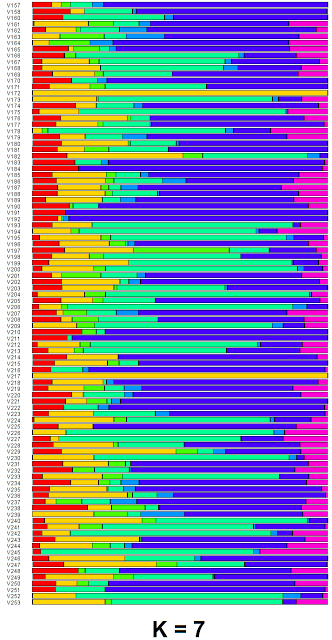
В целях сохранения совместимости с MDLP калькулятором, я остановился на модели, в которой выборка представлена в виде комбинации 7 предковых компонентов (K=7). Индивидуальные значения процентной составляющей каждого компонента в индивидуальном геноме (матрица Q), была визуализированы в R (ниже приведен график с результатами участников проекта MDLP, полный список доступен в этой таблице).
Полученные предковые компоненты (K=7) я обозначил следующими названиями (с сопутствующей цветовой легендой)**:
- Транс-кавказский — красный
- Балканском / средиземноморском -желтый
- Северо-кавказский -зеленый
- Западно-европейский
- Алтайский — светло-голубой
- Балто-славянский — темно-синий
- Прибалтийско-финский / Северо-европейский -фиолетовый
**Как обычно, названия компонентов условны и предназначены для мнемонических целей: исследователи должны быть осторожными при интерполяции предполагаемых компонентов в анализе этнической истории популяций.
На следующем этапе, я разбил все 371 индивидуальных «геномов» выборки на 22 фрагмента (каждый из которой соответствует аутосомной хромосоме) и затем использовал программное обеспечение Admixture для оценки структуры популяционного вклада в каждую из 22 хромосом. После этого я использовал пайплайн для перевода формата Plink в формат BEAGLE и последующего поэтапного преобразования фазированных данных BEAGLE обратно Plink формат.
Я предположил, что все образцы в моей выборке (представленной образцами VID) проекта MDLP возникли в в результате смешивание 7 отдельных предковых групп населения. Данное предположение означает, что «чистые» референсные группы населения тесно связаны с истинными предковыми популяциями. Исходя из этого предположения мы снова задействовали программное обеспечение Admixture, на этот раз с целью определения предковых компонентов в фазированном наборе данных из отдельных неполовых (аутосомных) хромосом.
Только после этой процедуры я смог использовать программу LAMP для определения уровня адмикса у отдельных индивидов. На практике, определение индивидуального уровня адмикса означает применение любой из указанных выше процедур, в которй используется либо модель «локус-специфического происхождения» (в случае, если предковые группы популяции априори неизвестны), либо модель «локус-специфического происхождения» гибридного населения. Затем полученные значения локус-специфического происхождения» отдельных сегментов в индивидуальном геноме усреднеяются и получаются значения долей адмикса в индивидуальном геноме.
Я расчитал в программном обеспечении Plink частоты аллелей (в стратифицированных по этническим признакам кластерах), и добавил в файл фиксированные частоты рекомбинации (определяются отдельно для каждой из 22 хромосом). Для моделирования динамического процесса смешивания предковых компонентов, я использовал различное количество поколений G ( 5, 10,25 поколений), предполагая 3 хронологически разных варианта, в которых при K = 7 предковые популяции A1, …, Ak, перемешивались в течение G = 5,10,25 поколений.
Результаты экспериментов для каждой из хромосом размещены в отдельные таблицы Excel, каждый из файлов Excel включает в себя следующие разделы:
1) результаты Admixture для фазированных генотипов хромосомы (Chr * -phased)
2) результаты Admixture для нефазированных генотипов хромосомы (Chr * -unphased)
3) результаты LAMP для G = 5 (Chr * -lamp-GEN5)
4) результаты LAMP для G = 10 (Chr * -lamp-GEN5)
5) результаты LAMP для G = 25 (Chr * -lamp-GEN5)
Образец этих выходных данных можно посмотреть в файле Excel с результатами анализа хромосомы 1 (Chr1).














Для отправки комментария необходимо войти на сайт.