Введение
Большинство из моих записей и экспериментов являются уникальными — именно по этой причине я очень часто дублирую эти записи на английском и русском языках. Без ложной скромности хочу представить читателю мое самое значимое достижение в области любительской персональной геномики — эксперимент по определению HLA серотипа на основе известных снипов из клиентских raw_data (файла с перечнем генотипированных снипов) компаний 23andme и FTDNA.
Однако перед тем как перейти к описанию самого эксперимента, необходимо вкратце напомнить о важности HLA-гаплотипов. Молекулярные различия между аллелями HLA могут варьироваться до 57 нуклеотидов в пептидо-связывающей кодирующего региона главный комплекса генов гистосовместимости (MHC) человека, однако еще до конца не установлено, являются ли эти молекулярные полиморфизмы результатом случайного стохастического процесса или же в процессе естественного отбора возникли селективные ограничения, связанные с функциональными различиями между молекулами HLA . Хотя HLA аллели, как правило, рассматривается в популяционно-генетических исследованиях в качестве эквидистантных молекулярных единиц, однако последовательность ДНК, и популяционное разнообразие также имеет решающее значение для интерпретации наблюдаемого полиморфизма HLA ( (Buhler S, Sanchez-Mazas A, 2011 HLA DNA Sequence Variation among Human Populations: Molecular Signatures of Demographic and Selective Events. PLoS ONE 6(2): e14643. doi:10.1371/journal.pone.0014643).
Стоит отметить еще один важный момент: HLA локусы характеризуются наиболее максимальным уровнем генетического разнообразия из всех человеческих генетических систем. Предварительные знания о степени разнообразия играет важную роль в эволюции и отборе молекулярных методов типирования. Определение частот аллелей также важны и в трансплантологии — например при аллогенетической трансплантации гематопоэтических стволовых клеток аллели используются для определения вероятности нахождения соответствующих «гистосовместимых» доноров для каждого пациента. Генетическое разнообразие локусов HLA отвечает также за эффективность работы иммунной системы при устранении клеток, несущих чужеродные антигены. Существует необходимость разработать методы оценки этого генетического разнообразия с целью изучения того, как различные группы людей реагируют на воздействие чужеродныъ антигенов, а также для оценки вклада каждого локуса HLA.
Система HLA была тщательно изучена также и с эволюционной точки зрения. Эта система содержит ряд тесно связанных генов, продукты экспрессии которых определяют множеством функций, связанных с регулированием иммунного ответа. Кроме того, последние исследования в области медицинской генетики установили в этом геномном регионе целый ряд вариантов, обуславливающих генетическую предрасположенность к более чем 40 заболеваниям. Ряд наблюдений показывает, что система человеческого HLA подвержена существенному влиянию естественного отбора, что приводит к появлению большого числа полиморфизмов с очень четко выраженным градиентом частот аллелей. Возникновение высокого уровня изменчивости в локусах имеет решающее значение для распознавания антигена, определения возраста аллелей и закономерностей в нарушении равновесного сцепления между локусами. Форма воздействия отбора неизвестна. Большинство исследователей склоняется к тому, что сложный характер наследования HLA вряд ли можно объяснить одним лишь воздействием естественного отбора. Мутации, рекомбинации и генетическая конверсия — все это также способствует увеличению изменчивости HLA. В то же время, очевидная древность многих аллелей HLA выявленных в ходе филогенетического анализа доказывают, что абсолютные темпы производства новых вариантов не являются высокоми. Детальное изучение популяционной и эволюционной особенности региона HLA, необходимо для обоснованного обсуждения эволюции генетических вариантов, предрасполагающих к определенным типам болезней (Thomson G.HLA population genetics.1991 Jun;5(2):247-60.).
Большинство из обычных людей сталкивались с термином антиген HLA только в процессе сдачи/получения донорских органов. В некоторых случаях имеются показания к выявлению типов антигенов HLA у родителей при осложнениях во время беременности:
При совпадении родителей по антигенам HLA увеличивается количество повторных аномальных беременностей с неустановленными причинами аномалий (Gerencer et al., 1978). Установлено отсутствие антигенов HLA в трофобласте, обнаруживается только b2-микроглобулин (Bodmer, 1981). Показано, что несовместимость по антигенам HLA матери и плода благоприятствует нормальному протеканию беременности и развитию плода.
Тот же самый авторитетный источник дает вполне сносное и вполне доступное для понимания начинающих любителей генетики:
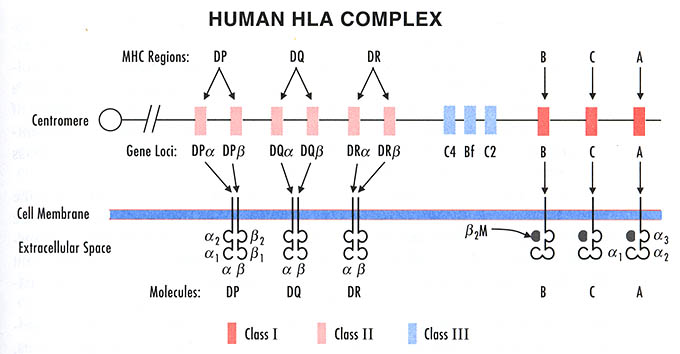
Главная система тканевой совместимости (major histocompatibility Complex — МНС) может быть рассмотрена в качестве еще одного примера комплекса тесно сцепленных локусов. МНС человека обозначается латинскими буквами HLA (human leukocyte antigene) и называется системой или комплексом HLA.
Годом открытия главной системы тканевой совместимости человека считают 1958 г., в котором Ж. Доссе открыл первый антиген, обозначенный Mac (современное обозначение — HLA-A2) (Снелл и др., 1979). С момента открытия первого антигена тканевой совместимости велась неуклонная работа по уточнению типирования тех или иных антигенных специфичностей, открытию новых антигенов, изучению их строения, разработке универсального метода типирования антигенов, локализации на хромосоме генов системы HLA, выяснению характера наследования, а также по совершенствованию антисывороток, выявляющих тканевые антигены и по выявлению особенностей распространения антигенов в различных популяциях мира.
Интенсивному развитию исследований способствовали в значительной мере Международные рабочие совещания по совместимости тканей. Первое такое совещание было организовано Д.Б. Эймосом в 1964 г. Затем были проведены рабочие совещания в 1965, 1967, 1970, 1972, 1975, 1977, 1980 и 1984 гг. Материалы Международных рабочих совещаний по совместимости тканей публикуются в качестве периодического издания “Histocompatibility Testing” и отражают основные этапы в развитии иммуногенетики человека и тканевого типирования. В 1967 г. после окончания очередного рабочего совещания был организован Международный номенклатурный комитет под руководством ВОЗ, который осуществляет разработку номенклатуры комплекса HLA, соответствующей уровню развития отрасли. Номенклатуры системы HLA выходили в Бюллетенях ВОЗ в 1968, 1972, 1975 и 1978 гг., а также в материалах Международных рабочих совещаний по совместимости тканей в 1970 и 1980 гг. Новая номенклатура была принята после окончания работы 9-го Международного рабочего совещания в 1984 г. (Bodmer et al., 1985). В таблице 1 номенклатура представлена для локусов HLA-A, -B, -C и -DR. Латинские буквы перед цифрами обозначают принадлежность к тому или иному локусу, а цифры — номер антигена. У некоторых специфичностей есть еще добавочное “w”, обозначающее, что данные специфичности либо не признаны к этому моменту всеми лабораториями, либо есть сложности в их типировании.
Каждое название специфического типа HLA или как еще говорят HLA аллелей имеет свой уникальный номер, соответствующий до четырех наборов цифр, разделенных двоеточиями. Длина обозначения аллели зависит от последовательности аллеля. Как правило, все аллели имеют по крайней мере четырех-символьное обозначения, которое соответствует двум наборам цифр, длинная номенклатура назначается только по необходимости. Цифры до первого двоеточия описывают тип, который практически всегда соответствует серологическому антигену, который несет данный аллотип. Следующий набор цифр, который используется в списке подтипов является обозначением порядка, в котором были определены последовательности ДНК. Аллели, номера которых отличаются двумя наборами цифр, отличаются друг от друга одной или несколькими нуклеотидными заменами которые ведут к изменению аминокислотной последовательности кодируемого белка. Аллели, которые отличаются только синонимичными/некодирующими нуклеотидными заменами в кодирующей последовательности, отличаются друг от друга использованием третьего дополнительного набора цифр. Аллели, которые отличаются только последовательностью полиморфизмов в интронах или в 5 ‘и 3’ нетранслируемых регионах, которые примыкают к экзонам и интронам, отличает использование четвертого набора цифр (см.дополнительную информацию).
Пример
HLA-A определяет HLA локуса
HLA-A1 серологическое обозначение антигена
HLA-A * звездочкой обозначается HLA аллелей определяется методами молекулярного типирования.
HLA-A * 01 2-разрядное разрешение обозначает группы аллелей, которые соответствует обычным серологическим группам — с низким разрешением
HLA-A * 0101 4-разрядное разрешение — последовательность различий между аллелями, которые приводят к аминокислотным заменами
HLA-A10101 60-значное разрешение — определяет некодирующие варианты, т.е последовательность изменений, не ведущих к аминокислотным заменам

В целях понимания уникальности проведенного мною эксперимента необходимо еще пояснить особенности определения типов антигенов. До прихода эра молекулярной геномики, антигены HLA подразделяются выявлялись серологически и с помощью лимфоцитов. К последним относятся антигены Dw, DQ, DP и некоторые DRw, относящиеся к области D. Большинство исследований фоксируется лишь на серологически определяемых антигенах, относящихся к локусам HLA-A, -B, -C и -DR. Первые три группы антигенов представляют собой гликопротеиды, обнаруживающиеся на клеточной мембране любой ядросодержащей клетки (Amos, Kostyu, 1980).
Как следует из вышесказанного, традиционно на протяжении десятилетий для HLA-типирования использовались серологические метода много десятилетий, и при этом серологическое типирование II класса молекул главного комплекса гистосовместимости человека зависело от адекватной экспресии этих молекул на поверхности В-лимфоцитов, наличия жизнеспособных клеток и полным набором иммунных сывороток. Тем не менее, применение молекулярно-генетических методов первого поколения (RFLP, PCR, SSO и т.д.) для типирования HLA привело к ситуации, в которой почти каждая лаборатория может независимо выполнять ДНК-типирование для определения аллеля HLA.
HLA типы и сцепленные SNP-ы на 6 хромосоме
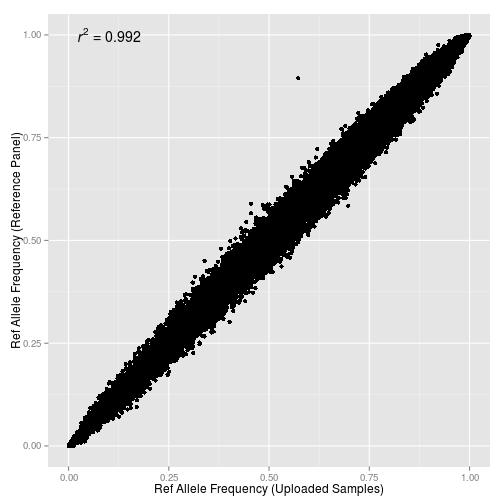
Как показал мой эксперимент, что даже того относительно небольшого количества снипов, которое типируется коммерческими компаниями в регионе MHC-HLA достаточно для определения типа антигена с высокой точностью.
К сожалению, имеется целый ряд нюансов значительно усложняющих анализ. Во-первых, определение классических аллелей HLA (например, HLA-, HLA-B и др.), с помощью новой чип-технологии, используемой в популярных коммерческих услугах геномных компаний (23andMe, FTDNA Family Finder и deCODEme), является очень сложным процессом, требующим проведения большого количества мультиплекс-ПЦР-реакций для получения полного генотипа пациента. Именно поэтому классические методы типирования HLA зачастую оказываются непрактичными в крупномасштабных исследованиях.Во-вторых, техническая сторона выявления типа HLA на основании генотипов является относительно сложной, и я не уверен что рядовой пользователь сможет ее осилить. При я могу помочь определить тип антигенов в качестве коммерческой услуги (которая примерно на 50 процентов дешевле стоимости стандартной медицинско-генетической услуги серотипирования).
К счастью для нас, в Центре генетики человека в Wellcome Trust Центра был разработан метод определения классических аллелей на основании анализа сцепленных SNP-ов. Метод был реализован в виде специального фреймворка (HLA * IMP) (Dilthey, A. T., Leslie, S., Moutsianas, L., Shen, J., Cox, C., Nelson, M. R., McVean, G. (2012): «Multi-population classical HLA type imputation» (submitted to Plos Comp Bio)).
HLA * IMP определяет HLA тип на основе данных о генотипе SNP, используя для этих итеративный метод отбора наиболее информативных полиморфизмов для отдельных поддерживаемых чипов генотипирования (Affymetrix 500K, 900K Affymetrix, Illumina 300K, 550K Illumina, Illumina 650K, Illumina 1M). Таким образом, HLA * IMP позволяет исследователям определить тип HLA в ходе импутирования геномных данные, собранных из нескольких доступных наборов SNP путем сопоставления импутируемых данных с референсными данными более 2500 индивидов европейского происхождения. Референсные данные содержат как SNP-варианты, так и классические аллели HLA типов.
Референсная панель которую я использую в целях импутации пропущенных значений, включает в себя:
1) так называемый The British Birth Cohort 1958 года включающем в себя SNP-ы Illumina 1,2 м и Affymetrix 6.0 (TheWellcome TheWellcome Trust Case Control Consortium, 2007) — 2420 индивидуальных образцов х 7733 SNP в расширенном регионе HLA.
2) выборку образцов CEU из HapMap (Международный консорциум HapMap, 2007) и CEPH CEU + дополнительные образцы (дde Bakker et al., 2006) — 92 образца х 7733 SNP-а перекрывающиеся со снипами The British Birth Cohort 1958)
Программа использует аутосомные гаплотипов состоящих из SNP -ов BC-195и CEU , и именно эти гаплотипы были задействованы в качестве эталонного набора данных. Была определена взаимосвязь типов HLA и гаплотипов SNP (эти гаплотипы были фазированы с помощью программы PHASE (Stevens and Scheet, 2005) с применением стандартных параметров фазирования многоаллельных локусов. Вместе с эталонным набором данных было получено около 5024 гаплотипов высокого разрешения, имеющими прямую корреляцию с 7733 SNP-ами в геномном регионе HLA. Эти гаплотипы определяют разное количества типов антигенов -2474 типа (HLA-A), 3090 (HLA-B), 2022 (HLA-C), 175 (HLA-DQA1), 2629 (HLA-DQB1), 2665 (HLA-DRB1). Это конкретные SNP-гаплотипы которые в дальнейшим программа использует для инференции типа антигена.
Эксперимент с выборкой проекта MDLP.
Поясню вкратце суть эксперимента
Как я уже упоминал в блоге MDLP (Re: Хромосома 6), геномные инструменты 23andme — RelativeFinder и AncestryFinder выделили целый кластер полуидентичных сегментных совпадений в который попало примерно 315 геномных совпадений с другими клиентами из базы данных 23andme. Примечательно, что все эти участки совпадения полностью или частчино перекрываются и расположены в одном и том же субрегионе области HLA-MHC на 6 хромосоме (21Mb-38MB). Эта замечательное скопление совпадающих участков составляет почти половину от общего количества моих AF / RF совпадений (315/720 или 43,75%).
Ранее я предположил, что столь явный перекос числа общих по происхождению полуидентичных сегментов в области HLA свидетельствует о том, что один из гаплотипов совпаденцев является идентичным типом антигенов HLA. До недавнего времени, мое предложение опиралась исключительно на мои интуитивные догадки. Затем я смог найти ключ решения проблемы с помощью HLA * IMP и до сих пор, после чего мне удалось провести эксперимент в HLA * IMP , в ходе которого я применил методологию к анализу данных 23andMe (Illumina Omnio Express) .
Условия эксперимента
Для успешного проведения моих тестов, я должен был убедиться, что мои собственные данные соответствуют следующим требованиям:
* SNP-ы должны входить в область xMHC ( на 6 хромосоме )
* Отобранные в первоначальной выборки кандидаты должны быть европейского происхождения
* Высокое качество и плотность типированных SNP в регионе HLA, что является критическим условием повышения точности импутации
* Поскольку HLA IMP не обеспечивает прямой поддержки кастомных модифицированных чипсетов 23andMe, и я был ограничен в своем использовании комбинированным набором генотипов с двух чиспетов 23andMe (v2 и v3), то мне пришлось «понизить» число SNP-ов в версии платформы Illumina (Illumina 300K). Но и этого оказалось достаточно.
Тест гипотезы
Для того, чтобы проверить свое первоначальное предположение о том что вышеупомянутые совпаденцы разделяют одинаковый гаплотип HLA, я выбрал 7 участников из cвоих проектов (себя, свою мать; человека, о котором заведомо было известно, что у него есть наполовину идентичный совпадющий сегмент со мною и моей матерью в xMHC область; oстальные участники использовались в качестве контрольной группы).
C этой целью я преобразовал исходные данные 23andMe участников проекта в формат Plink, затем объединил файлы в один набор данных, и выделил подмножество SNPs на 6-ой хромосоме , используя команду Plink — сhr 6. После этого я преобразовал файл с данными генотипов из формата Plink во входной формат данных HLA * IMP. В качестве следующего шага, я провел контроль качества данных путем удаления SNP-ов и частных лиц со слишком большим количеством отсутствующих данных, а также привел в соответствие (за счет выравнивания) дополнительные SNP-ы из референсной панели HapMap. Наконец, я поэтапно профазировал генотипы для получения гаплотипов Примечание: я также заменил ID задействованных участников проекта на префикс N.
Гаплотипные данные были затем загружены на серевер HLA * IMP, где и была произведена процедура импутации HLA типов.
Выявленные в ходе этой операции типы HLA выглядили следующим образом (каждый из индивидов представлен 2 гаплотипами, один из которых был унаследован от матери, другой от отца): HLA-A: HLA-B: HLA-C: HLA-DQA: HLA-DQB: HLA-DRB.
| IndividualID |
Chromosome |
HLAA |
HLAB |
HLAC |
HLADQA |
HLADQB |
HLADRB |
| N1 |
1 |
101 |
801 |
701 |
501 |
201 |
301 |
| N1 |
2 |
2601 |
2705 |
102 |
101 |
501 |
101 |
| N6 |
1 |
3101 |
801 |
701 |
501 |
201 |
301 |
| N6 |
2 |
201 |
1501 |
304 |
501 |
201 |
301 |
| N3 |
1 |
6801 |
1501 |
102 |
101 |
501 |
101 |
| N3 |
2 |
2301 |
5201 |
501 |
101 |
501 |
101 |
| N2 |
1 |
101 |
801 |
701 |
501 |
201 |
301 |
| N2 |
2 |
2601 |
3801 |
1203 |
102 |
602 |
1501 |
| N5 |
1 |
301 |
1501 |
304 |
501 |
302 |
401 |
| N5 |
2 |
205 |
5001 |
602 |
501 |
202 |
701 |
| N7 |
1 |
101 |
801 |
701 |
501 |
301 |
1101 |
| N7 |
2 |
101 |
1501 |
303 |
103 |
604 |
1301 |
| N4 |
1 |
301 |
702 |
702 |
401 |
402 |
801 |
| N4 |
2 |
2402 |
4002 |
202 |
501 |
301 |
1101 |
|
|
Гаплотипы в приведенной выше записи следует читать следующим образом (например, в случае N1): HLA * 0101: Cw * 0701: B * 0801: DRB1 * 0301: DQA1 * 0501: DQB1 * 0201.
В вышеприведенной таблице можно отметить совпадение одного из гаплотипов у участников эксперимента под номерами N1, N2 и N7, т.е они имеют идентичный гаплотип.
Это подтверждает одну из моих рабочих гипотез. У индивидов N1 (моей матери), N2 (меня) и N7 с помощью инструмента Relative Finder от 23andme в геномном регионе HLA на 6 хромосоме был задетектирован полуидентичный сегмент ДНК, ( этот сигмент отвечает формальным требованиям идентичного по происхождению сегмента, то есть генетическая дистанция превышает порогое значение в 7 сантиморганид (сокращенно сМ — это единица измерения вероятности кроссинговера между двумя генами) и число снипов в непрервыном полуидентичном сегменте равно 700 ).
Таким образом, можно с уверенностью утверждать, что мое первоначальное предположение, подкрепляется результатами установления типа HLA.
Практические результаты испытаний
Практически каждый из известных и описанных в литературе типов человеческих HLA имеет убедительную связь с так называемыми медицинскими рисками. Мы не будем сейчас останавливаться на них, и отложим рассмотрение этой тематики на будущей. Однако помимо медицинской полезности, существуют также и некоторые преимущества знания своего типа HLA и с точки зрения генетической генеалогии:
1) Прежде всего, это возможность определить характер распространения сегментов в области xMHC на хромосоме 6. Приведу конкретный пример на своих собственных данных — вышеупомянутый «расширенный» гаплотип HLA * 0101: Cw * 0701: B * 0801: DRB1 * 0301: DQA1 * 0501: DQB1 * 0201 (в англоязычной литературе встречаются и другие обозначения —AH8.1, COX,[1] Super B8, ancestral MHC 8.1[2] or 8.1 ancestral haplotype; далее мы будем писать его в сокращенном виде: A1 :: DQ2). По мнению некоторых исследователей этот гаплотип A1 :: DQ2 представляет собой загадку с точки зрения изучения эволюционного процесса рекомбинаци. Дело в том, что скорость рекомбинации этого гаплотипв A1 :: DQ2 не соответствует теоретическими ожиданиям в плане скорости рекомбинации. Нам известно, что в регионах Европы, где этот гаплотип впервые сформировался и начал распространятся, существуют другие гаплотипы, некоторые из которых являются «предковыми», обладая при этом весьма значимой длиной. Однако рассматриваемый гаплотип является своего рода рекордсменом — он включает в себя последовательный контиг ДНК длинной примерно в 4,7 миллионов (!) пар нуклеотидов, и около 300 генных локусов. Кроме того, все исследования отмечают высокую «резистентность» этого гаплотипа по отношению к рекомбинации. В качестве объяснения этого уникального феномена выдвигаются разные гипотезы — некоторые полагают что обструкция рекомбинации в этом гаплотипе была неким неизвестным была жестким образом кодирована в самой ДНК (по аналогии с хардкодингом -жестким «вшиванием» в программный код различных данных, касающихся окружения программы) Другие объясняют эту аномалию последствиями многократного селективного отбора гаплотипа. Здесь следует прояснить суть проблемы: согласно классической теории рекомбинации, физическая длина любого аутосомного гаплотипа (то есть число нуклеотидов входящих в него) из-за быстрых темпов эволюционной рекомбинации должна неизменно уменьшатся с каждым поколением. Однако гаплотип A1 :: DQ2 является живым опровержением универсальности этого представления.
2) Во-вторых, можно попытатся произвести оценку времени и места возникновения этого гаплотипа. В ходе систематического обследования мировых популяций, регулярно проводимого сетью центров и институтов трансплантологии, было установлено, что гаплотип A1 :: DQ2 является наиболее часто встречается у белых жителей США , ~ 15% которых имеет этот гаплотип. Анализ SNP-ов сцепленных с этим типом, дает основания полагать, что гаплотип мог возникнуть примерно 20.000 лет в Европе, хотя эта интерпретация в настоящее время признается некоторыми исследователями неубедительной. Согласно этой интепретации, гаплотип мог возникнуть в результате климатических изменений во врема последнего ледникового максимума примерно 11500 лет назад (поэтому этот гаплотип называют иногда предковым европейским гаплотипом, или гаплотипом A1-B8 (AH8.1). Этот один из тех 4 гаплотипов HLA, которые являются общими для западных европейцев и азиатов. Если предположить, что частота распространения этого гаплотипа в начальной популяции составляла 50% во время последнего ледникового максимума и плавно снижалась на 50% каждые 500 лет то частота гаплотипа в современных популяциях не должна превышать 0,1% в любой европейской популяции. Однако реальная частота составляет, как было показано выше, примерно 10%, что превышает ожидаемую в теории частоту распространения почти в 100 раз. Применительно к генетической генеалогии, это подробное разъяснение может означать только то, что массовое скопление совпадающих сегментов в xMHC регионе на 6 хромосоме может быть четким указанием на наличие очень далеких общих предков (вплоть до жителей эпохи неолита). При любом раскладе, предлагаемый RelativeFinder/23andme интервал в 5-10 поколений до последних общего предка для региона xMHC является нереалистично заниженным.
2) Кроме того, используя стандартные средства геногеографии можно сделать вывод о географическом ареале распространения конкретного гаплотипа HLA. Опять-таки, изучая геногеографию все того же гаплотипа A1 :: DQ2, можно увидеть, что его локальные пики приходятся на весьма удаленные друг от друга регионы — это Исландия, регион исторического проживания поморов на Севере России, северная Сербия, земля басков, а также те регионы Мексики, которые массовао заселялись басками. Общей чертой этих регионов является не географическая близость, а крайняя степень изоляции.
Относительно высокая частота распространения гаплотипа в наиболее изолированных географических регионах Западной Европы, Ирландии, Скандинавии и Швейцарии наряду с крайне низкой частотой во Франции и латинизированной части Иберии является результатом описанной в популяционной генетике и антропологии модели замещения, народонаселения Европы, которое обычно связывается с началом эры неолита. В этом случае возраст рассматриваемого гаплотипа в Европе, превышает 8000 лет.
Напоследок хочу порекомендовать —The Allele Frequency Net Database -хорошую базу данных по частотам аллелей полиморфичных регионов HLA,KIR,Cytokine,MIC Института транплантной иммунологии при Ливерпульском университете. Она является очень удобным инструментом для анализа частот HLA гаплотипов в мировом масштабе.
В частности, можно производить поиски по гаплотипам, и получать на выходе данные о частотах в различных популяциях, а также визуализировать полученные данные на карте.
Пример (гаплотип A1 :: DQ2)
| 1 |
A*01:01—B*08:01—C*07:01—DRB1*03:01—DQB1*02:01 |
 |
Ireland South |
11.50
|
 |
250
|
|
| 2 |
A*01:01—B*08:01—C*07:01—DRB1*03:01:01-DQB1*02:01 |
 |
England North West |
9.50
|
 |
298
|
|
| 3 |
A*01:01—B*08:01—C*07:01—DRB1*03:01—DQB1*02:01-DPB1*04:01 |
 |
Ireland South |
8.30
|
 |
250
|
|
| 4 |
A*01:01—B*08:01—C*07:01—DRB1*03:01—DQB1*02:01 |
 |
Poland |
4.00
|
 |
200
|
|
| 5 |
A*01:01—B*08:01—C*07:01—DRB1*03:01—DQB1*02:01 |
 |
USA Hispanic pop 2 |
1.78
|
 |
1,999
|
|
| 6 |
A*01:01—B*08:01—C*07:01—DRB1*03:01—DQB1*02:01-DPB1*01:01 |
 |
Ireland South |
1.40
|
 |
250
|
|
| 7 |
A*01:01—B*08:01—C*07:01—DRB1*03:01—DQB1*02:01 |
 |
USA African American pop 4 |
1.39
|
 |
2,411
|
|
| 8 |
A*01:01—B*08:01—C*07:01—DRB1*03:01—DQB1*02:01 |
 |
USA Asian pop 2 |
0.09
|
 |
1,772
|
|














Для отправки комментария необходимо войти на сайт.