Как я уже отмечал в своих предыдущих записях, за последние годы был опубликован ряд работ, посвященных попыткам генотипирования останков древних людей — от живших несколько тысячелетий назад до «усть-ишимца» с предположительным возрастом около 45 тысяч лет, неандертальцев и «денисовки». Количество таких расшифровок растет все быстрее, что не может не вызывать оптимизма. Вторая половина 2014 года особенно примечательна как количеством подобных публикаций, так и числом полных геномных NGS-сиквенсов древних людей, размещенных в публичных репозиториях (банках геномных данных). Так, в сентябре в Nature была опубликована окончательная версия работы Lazaridis et al. 2014 «Ancient human genomes suggest three ancestral populations for present-day Europeans». Работа получила широкое освещение в СМИ, поскольку аналитическая выборка сэмплов в этом исследовании включала значительное количествао заново генотипированных (на чипе Affymetrix HumanOrigin) образцов ДНК из древних палеолитических стоянок Сибири (Афонтова Гора, Малта), представителя древней индейской культуры Кловис и палеоэскимоса Cаккак. В работе был представлен целый ряд образцов древней ДНК представителей европейских мезолитических и неолитических культур, опубликованных в более ранних работах 2012-2014 годов: Skoglund et a. 2014 «Genomic Diversity and Admixture Differs for Stone-Age Scandinavian Foragers and Farmers»(шведские земледельцы и охотники собиратели эпохи неолита); Olalde et al. 2014 «Derived immune and ancestral pigmentation alleles in a 7,000-year-old Mesolithic European» (дДНК мезолитического населения Иберийского полуострова) и т.д.
В этой связи необходимо также отметить статью Carpenter et al. 2013 «Pulling out the 1%: whole-genome capture for the targeted enrichment of ancient DNA sequencing libraries», в которой целый авторский коллектив представил результаты исследований древних образцов ДНК найденных в захоронениях бронзового века II тыс. д.н.э (Болгария и Дания). В следующей работе опубликованной в конце октября, Gamba et al. 2014. «Genome flux and stasis in a five millennium transect of European prehistory», читателям была представлена хронологическая перспектива на процесс изменения генофонда населения популяций живших на территории Паннонской равнины на протяжении 5000 лет (с эпохи неолита до конца железного века), проиллюстрированная на примере изучения 13 образцов древней ДНК. Параллельно вместе с этим Wellcome Trust Sanger Institute разместил геномные «риды» геномов древних англосаксов и бриттов (сама статья еще находится в процессе пре-публикации, презентация статьи была представлена на последней конференции AJHG).
Более важные публикации появилась совсем недавно. В частности, таковой публикацией является статья Fu et al. 2014 «Genome sequence of a 45,000-year-old modern human from western Siberia» о геноме так называемого «усть-ишимца» (возраст останков которого датируются 45 000 д.н.э) и статья Seguin-Orlando et al. 2014 «Genomic structure in Europeans dating back at least 36,200 years», посвященная обсуждению результатов анализа ДНК знаменитого «папусоида» с палеолитической стоянки Костенки-14.Тело мужчины, жившего 37 тыс. лет назад и найденное в 1954 г. на юго-западе России, оказалось источником старейшей европейской ДНК. Анализ его генома, опубликованный на прошлой неделе, показывает, что большинство разнообразных европейских генетических комбинаций существуют более 30 тыс.лет и пережили последний ледниковый период. Генетики обнаружили что ДНК Костенки-14 является близкородственным по отношению к раннеевропейским охотникам-собирателям, современным европейцам и жителям Сибири.
В то же время другой древний геном, данные о котором были опубликованы несколько недель назад, принадлежащий сорокапятитысячелетнему западному сибиряку, известному как Усть-Ишим, имел родство как с европейцами, так и с азиатами. Любопытно, что в этой статье подтверждается то о чем я говорил гораздо раньше: процент неандертальских генов у древних евразийцев был выше чем у современных (о чем я упоминал в одной из своих заметок в этом блоге).
Трудами известного геномного блоггера Феликса Чандракумара большинство из них было переведено в простой и доступный формат, аналогичный файлам raw data от FTDNA и 23andMe. В GEDMatch можно поиграть с этнокалькуляторами и даже попытаться сравнить свой геном с геномами древних людей.Для этого следует взять из таблицы (кот. видна, если пройти по ссылке) номера, которыми обозначены древние геномы.
| Sample Name | Sample Location | GEDMatch | Sex | Y-DNA | Mt-DNA | Approx. Age by authors | My Analysis or Comments |
|---|---|---|---|---|---|---|---|
| Altai Neanderthal | Denisova Cave, Siberia | F999902 | Female | 50,000 years | |||
| Denisova | Denisova Cave, Siberia | F999903 | Female | 30,000 years | |||
| Palaeo-Eskimo | Qeqertarsuaq, Greenland | F999906 | Male | Q1a | D2a1 | 4,000 years | Palaeo-Eskimo 2000 BC DNA |
| Clovis-Anzick-1 | Montana, North America | F999919 | Male | Q-Z780 | D4h3a | 12,500 years | Matches Living people. |
| Mal’ta | South-Central Siberia | F999914 | Male | R | U | 24,000 years | Matches Living people on X Chromosome. |
| La Braña-Arintero | León, Spain | F999915 | Male | C-V183 | U5b2c1 | 7,000 years | Analyzing La Braña-Arintero Ancient DNA |
| Motala-12 | Östergötland, Sweden | F999917 | Male | I-L460 | U2e1 | 7,000 years | My Analysis of Motala-12 ancient DNA |
| LBK | Stuttgart, Germany | F999916 | Female | T2c2 | 7,500 years | Matches Living people | |
| Loschbour | Loschbour, Luxembourg | F999918 | Male | I-L460 | U5b1a | 8,000 years | Matches Living people |
| Ajvide58 | Sweden | F999924 | Male | I-CTS772 | U4d | 5000 years | Ajvide58 DNA Analysis |
| Gökhem2 | Sweden | F999934 | Female | H1c | 5000 years | Gökhem2 Ancient DNA Analysis | |
| Hinxton-2 | Cambridgshire, UK | F999921 | Female | H2a2b1 | 1300 years | Hinxton-2 Analysis | |
| Hinxton-3 | Cambridgshire, UK | F999922 | Female | K1a4a1a2b | 1300 years | Hinxton-3 Analysis | |
| Hinxton-4 | Cambridgshire, UK | F999925 | Male | R-DF25 | H1ag1 | 2000 years | Hinxton-4 has X-Matches with living people |
| Hinxton-5 | Cambridgshire, UK | F999926 | Female | H2a2a1 | 1300 years | Hinxton5 Ancient DNA Analysis | |
| KO1 | Tiszaszőlős-Domaháza, Hungary | F999931 | Male | I-L68 | R3 | 5650-5780 cal BC | Analysis of Neolithic KO1 genome |
| NE1 | Polgár-Ferenci-hát, Hungary | F999937 | Female | U5b2c | 5070-5310 cal BC | NE1 Ancient DNA Analysis | |
| NE5 | Kompolt-Kigyósér, Hungary | F999927 | Male | C-F3393 | J1c | 4990-5210 cal BC | Ancient Hungarian Genome NE5 Analysis |
| NE6 | Apc-Berekalja I., Hungary | F999932 | Male | C-P255 | K1a3a3 | 4950-5300 cal BC | Analysis of Hungarian genome-NE6 |
| NE7 | Apc-Berekalja I., Hungary | F999928 | Male | I-L1228 | N1a | 4360-4490 cal BC | Ancient Hungarian genome — NE7 |
| CO1 | Apc-Berekalja I., Hungary | F999930 | Female | H | 2700-2900 cal BC | Analysis of Copper age genome CO1 | |
| BR2 | Ludas-Varjú-dűlő, Hungary | F999933 | Male | J-M67 | K1a1a | 1110-1270 cal BC | Ancient BR2 matches living people |
| IR1 | Ludas-Varjú-dűlő, Hungary | F999929 | Male | N-M231 | G2a1 | 830-980 cal BC | Ancient Hungarian genome — IR1 |
| Tyrolean Iceman (ERP001144) |
Tisenjoch Pass, Oetztal Alps | Male | 5300 years | Pending | |||
| Ust’-Ishim | Ust’-Ishim, Siberia | F999935 | Male | K-M526 | R | 45,000 years | Ust’-Ishim matches with living people! |
| Kostenki14 | European Russia | F999936 | Male | C-V199 | U2b | 38,700-36,200 years | Kostenki14 Ancient DNA Analysis |
| Sample Name | Sample Location | Sex | Y-DNA | Mt-DNA | Approx. Age by authors |
|---|---|---|---|---|---|
| Mezmaiskaya Neanderthal | Mezmaiskaya Cave | Female | 29,000 years | ||
| Tianyuan | Tianyuan Cave, China | R | 40,000 years | ||
| Afontova Gora-2 | South-Central Siberia | Male | R1? | R | 17,000 years |
| Motala-1 | Östergötland, Sweden | Female | U5a1 | 7,000 years | |
| Motala-9 | Östergötland, Sweden | Female | U5a2 or U5a1f1a1 | 7,000 years | |
| Motala-6 | Östergötland, Sweden | Male | U5a2d | 7,000 years | |
| Motala-2 | Östergötland, Sweden | Male | F-P139 | U5e1 | 7,000 years |
| Motala-4 | Östergötland, Sweden | Female | U5a2d | 7,000 years | |
| Motala-3 | Östergötland, Sweden | Male | I-M258 | U2e1 | 7,000 years |
| Hinxton-1 | Cambridgshire, UK | Male | R-L151 | K1a1b1b | 2000 years |
| Ajvide53 | Sweden | Female | U4d | 5000 years | |
| Ajvide59 | Sweden | Male | I-PF3796 | U5b2c1 | 5000 years |
| Gökhem7 | Sweden | Female | H | 5000 years | |
| Ire8 | Sweden | Male | I-CTS6343 | U4d | 5000 years |
| StoraFörvar11 | Stora Karlsö, Sweden | Male | I-CTS4077 | U5a1f1a | 7500 years |
| Gökhem4 | Sweden | Male | CF-M3690 | H | 5000 years |
| Gökhem5 | Sweden | Female | K1e | 5000 years | |
| Ajvide52 | Sweden | Male | HIJK-F929 | HV0a | 5000 years |
| Ajvide70 | Sweden | Female | U4d | 5000 years | |
| NE4 | Polgár-Ferenci-hát, Hungary | Female | J1c | 5050-5290 cal BC | |
| NE3 | Garadna, Hungary | Female | X2b | 5010-5210 cal BC | |
| BR1 | Kompolt-Kigyósér, Hungary | Female | K1c1 | 1980-2190 cal BC | |
| KO2 | Berettyóújfalu-Morotva-liget, Hungary | Female | K1 | 5570-5710 cal BC | |
| NE2 | Debrecen Tócópart Erdõalja, Hungary | Female | HV | 5060-5290 cal BC | |
| V2 | Vratitsa, Bulgaria | Male | U2e1’2’3 | 1500-1100 BC | |
| M4 | Borum Eshøj, Denmark | Male | B2 | 1350 BC | |
| K8 | Krushare, Bulgaria | Male | R | 450-400 BC | |
| NA43 | Laguna de los Condores, Peru | Male | B4b’d’e | 1000-1500 AD | |
| AusAboriginal | Western Austalian | Male | F-M235 | O1a | 100 years |
| NA41 | Laguna de los Condores, Peru | Male | L3 | 1000-1500 AD | |
| P192-1 | Svilengrad, Bulgaria | Male | U3b | 800-500 BC | |
| T2G2 | Stambolovo, Bulgaria | Male | H1c9a | 850-700 BC | |
| NA42 | Laguna de los Condores, Peru | Male | D1 | 1000-1500 AD | |
| NA50 | Laguna de los Condores, Peru | B4b’d’e | 1000-1500 AD | ||
| NA47 | Laguna de los Condores, Peru | L3 | 1000-1500 AD | ||
| NA40 | Laguna de los Condores, Peru | L3 | 1000-1500 AD | ||
| NA39 | Laguna de los Condores, Peru | Male | B2 | 1000-1500 AD | |
| Feld1 Neanderthal | Neander Valley, Germany | 42,000 years | |||
| Sid1253 Neanderthal | El Sidron cave, Asturias, Spain | 49,000 years | |||
| Vi33.16 Neanderthal | Vindija cave, Croatia | Female | 38,310 years | ||
| Vi33.25 Neanderthal | Vindija cave, Croatia | Female | |||
| Vi33.26 Neanderthal | Vindija cave, Croatia | Female | 44,450 years |
В своем блоге Феликс размещает аналитические отчеты по каждому из проведенных анализов, отчеты включают графическое отображения «состава различных геномных компонентов происхождения» каждого из образцов в калькуляторах Gedmatch (включая мой последний калькулятор K23b), фенотипические признаки (предположительный цвет кожи и глаз), возраст на момент смерти и т.д.
Пытаясь ответить на вопрос, насколько правдоподобны (в смысле реального генеалогического родства) результаты совпадения сегментов древних и современных людей, Феликс приводит замечательные вычисления оценки правдоподобия совпадений в геномах современных людей и древних образцов. К сожалению, рассуждения замечательные, но вызывающие определенные вопросы, которые я озвучу в другой заметке.
Так или иначе, поставленная Феликсом на поток и практически полностью автоматизированная работа с древними геномами заслуживает безусловного признания, поскольку в силу разделения труда позволяет другими исследователям-любителям полностью сконцетрировать свое внимание на процессе непосредственного анализа полученных данных, вместо того чтобы тратить свои ресурсы на процесс извлечения снипов из «сырых» геномных данных. Благодаря этому разделению труда, Давид Веселовский из проекта Eurogenes провел ряд замечательных экспериментов с этими данными (включая PCA, Treemix и вычисление генного дрейфа с помощью f3). В основном выводы этих экспериментов повторят то, что было написано в статьях профильных генетиков, за исключением одного интересного вывода на основании графа Treemix, в котором отображено направление процессов обмена генами между различными древними популяциями:
«В отношении Kostenki14, графики Treemix подтверждают один из основных выводов работы Seguin-Orlando et al. 2014, согласно которой главны компонент образца Kostenki-14 является базальным «предковым» компонентом более поздних европейцев (Basal_Eurasian). Тем не менее, два последних графика показывают, что этот базальный «компонент» не тот же самый «базальный» компонент в геноме неолитического образца из Штутгарта, связанного с базальным евразийским компонентом, который был описан в работе Lazaridis et al. 2013″.
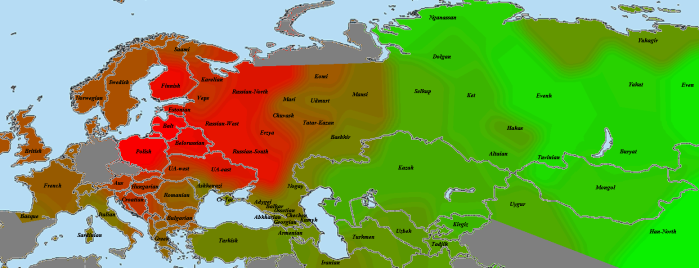
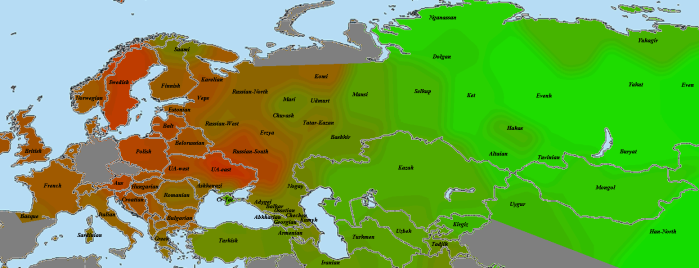
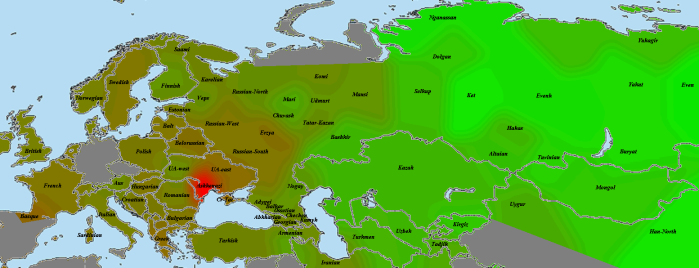
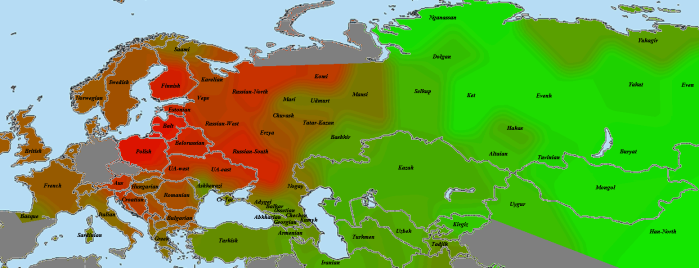
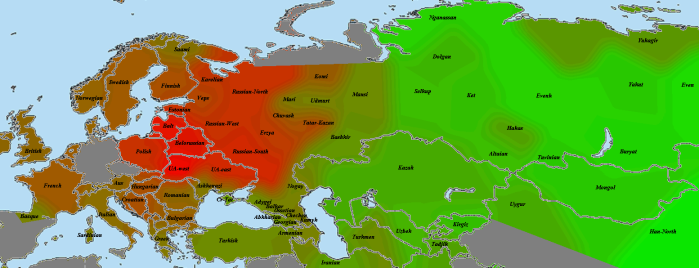
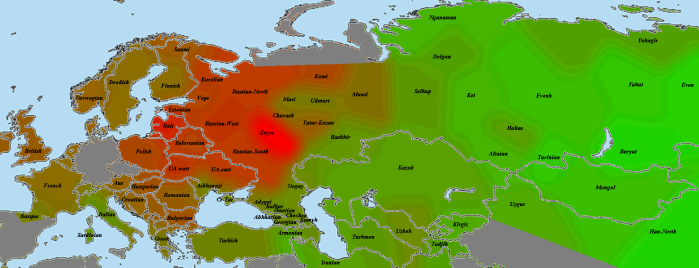
Другой геномный блоггер, Сергей Козлов, использовал те же самые данные палеогеномов (взятые с сайта Ф. Чандракумара) для создания замечательных карт, иллюстрирующих количество и интенсивность общих IBD-сегментов палеогеномов и геномов современных популяций.
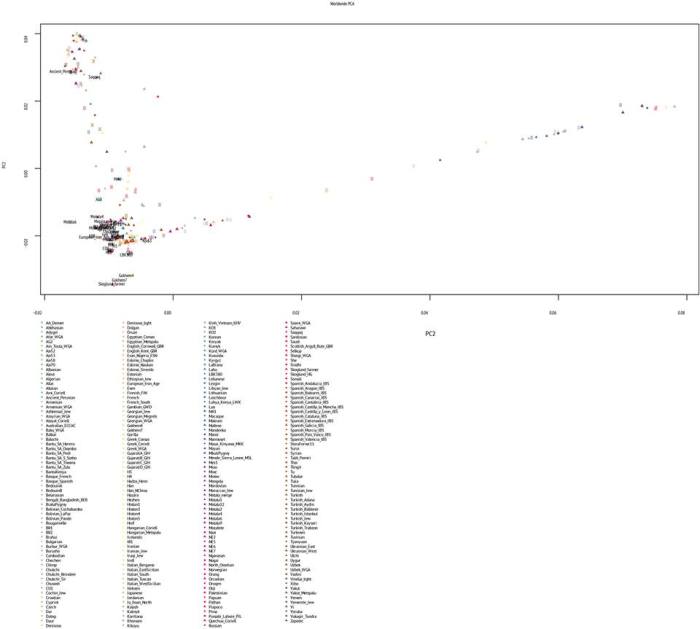
Я решил не оставаться в стороне и провел собственный анализ PCA и кластеризации популяций по значениям компонентов генетического разнообразия.
Ниже приведены иллюстрации к моему опыту кластеризации собственного генома с геномами древних жителей Евразии. В качестве входных данных алгоритма ward-кластеризации в программе R, я использовал собственные значения 4 векторов главных компонентов (PC) разнообразия. Эти векторы, в свою очередь, были получены путем вычислений в большом массиве (2024 образца) генетических данных (примерно 110 тысяч снип-полиморфизмов) представителей современных и древних популяций. Мой геном (обозначенный как Vadim) представляет собой набор, полученныq в ходе импутации по датасету Human Origin значения снипов информативных с точки зрения эволюционного происхождения, и используется в качестве контрольной группы.
Для начала график PCA, и положение палеогеномов на этом графике.
В аналитической выборке я задействовал снипы геномов высших и низших приматов (дендрограмма выборка укоренена на геноме мармозетки), древних гоминидов (денисовского человека и неандертальцев). Остальное — как я и упоминал выше — представляет собой совокупность снипов современных и древних популяций.
Благодаря характеру выборки и характеру используемых снипов, я могу взглянуть на свое происхождение с наиболее широкой перспективы, позволяющей проследить индивидуальный эволюционный путь от древнейших людей до наших современников.
Можно сказать, что я проделал самое далекое (из всех предыдущих) генеалогическое путешествие в собственное прошлое. Разумеется, без предыдущего выделения обработки образцов древнего ДНК новейшими биохимическими методами, а также публикации данных — это путешствие длинной в сотни тысяч лет не могло бы просто состоятся. Так что огромное спасибо всем биохимикам, генетикам и биоинформатиков работавшим с образцами древней ДНК.
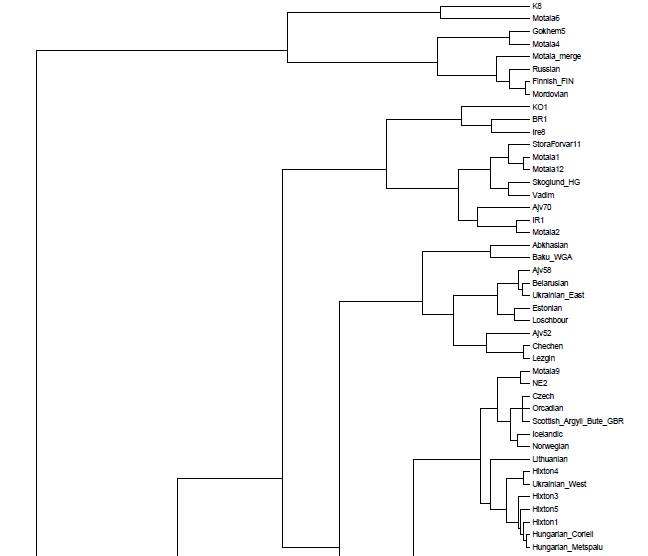
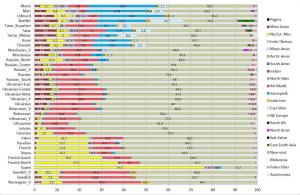
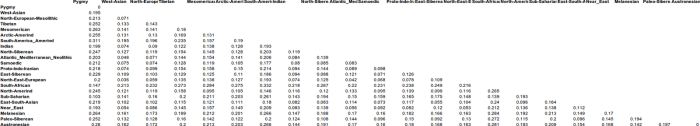
Полученные мной кластерные дендрограммы вышли очень большого разрешения. В силу этого, имеет смысл изучить топологию, структуры и расположение популяционных групп-кластеров в полномасштабном варианте, иначе могут возникнуть интересные вопросы.
Поэтому — я подготовил соответствующие файлы PDF и разместил ссылки на эти файлы для удобного просмотра.
Здесь их опубликовать не представляется возможным, и по этой причине я ограничу себя размещением тех фрагментов трех вариантов кластерных диаграмм, на которых присутствуют древние образцы.
Забегая вперед, можно заметить, что образцы ДНК древних людей (т.е людей современного анатомического типа — homo sapiens sapiens), строго говоря, разбиваются на три органические суперкластера — древних сибириков (или евразийцев), древних европейских охотников-собирателей, и ранних неолитических европейских земледельцев. В основной своей части состав и топология популяционных кластеров стабилен в разных вариантах, наибольшие видоизменения заметны у тех образцов, чье множество снипов имеет меньшее пересечение с общим набором снипов. Отсюда довольно таки тривиальный вывод: чем меньше общее число снипов — тем больше флуктуаций наблюдается в расположении древних образцов внутри ветвей кластерной дендрограммы.
Кластер древних евразийцев наиболее стабилен (т.к. там всего два древних генома Afontova Gora 2 (AG2) и знаменитый мальчик с сибирской стоянки Malta (MA1); причем оба образца взяты из одного источника данных). Из современных популяций к этому кластеру наиболее органично примыкают различные группы населения центральной Азии — от таджиков до гуджаратов, и от калашей до пуштунов.
Кластер древних охотников-собирателей Европы наиболее неустойчив, и это объясняется прежде всего разным числом снипов в образцах, а также тем что сами образцы взяты из разных исследований. Тем не менее тенденция наглядна — древнейшие европейцы (охотники-собиратели мезолита) наиболее близки по своим аутосомным снипам к жителям современной западной и северной Европы — особенно Британских островов, Скандинавии и Балтийского региона. Практически во всех вариантах прибалтийцы близки к древним жителям Швеции (Готланда), а также мезолитическим образцам La Brana, Motala и Loshbour. Последние также близки к финнам, эстонцам и северным русским. Из более поздних и географически удаленных образцов к ним близки древние образцы из Венгрии неолитического периода, бронозового и железного веков (BR1, NE2 и KO1).
Интересно, что в этот же кластер входят как современные популяции западной Европы (британцы, норвежцы, французы и др.), так и современные жители центральной Европы — чехи хорваты и венгры. Является ли это наследием древних времен (гальштатской общности связываемой с древними кельтами) — трудно сказать. Не этим ли объясняется тот факт, что образцы древних англо-саксов и бриттов (обозначенные здесь как Hixton) иногда кластеризируются с (современными!) венграми, хорватами, иногда с современными англичанами из Кента и корнуэлльцами. При этом некоторые из образцов Hixton остаются близки (в смысле схожести генома) к скандинавам, оркнейцам, шотландцам, и даже литовцам.
Мой собственный «геном» (Vadim) также входит в эту группу, причем в разных вариантах он определенно близок одновременно и древним мезолитическим и эпинеолитическим шведам, а также более поздним образцам из Венгрии (киммерийского мальчика IR1, а также самый «балтийский» из всех древних венгерских обрацов — KO1). Интересно что IR1 («аутосомный геном» «киммерийского » мальчика Y-гаплогрупы N1a из захоронения паннонской культуры бронзового века Mezőcsát примерно 900 год до нашей эры) в первых четырех главных компонентах кластеризируется с моим собственным «аутосомным геномом»). Это наверное объясняет почему мой собственный геном дает хорошие комбинации (fit) к комбинации трапезундских турков и древних жителей Балтийского региона.
Как известно, попгенетики готовят к публикации большую статью, в которой подводятся итоги нескольких лет исследования генофонда представителей древних культуры шнуровой керамики* (известной также как культура боевых топоров) и ямной культуры** (другое название — древнеямная культурно-историческая общность). Безусловно, это исследование обещает пролить свет на некоторые темные места генетических связей жителей этих культур с современным населением Восточной Европы (особенно Польши, Украины, Беларуси и юго-западной части России).
Пока все детали исследования неизвестны, однако благодаря настойчивости некоторых энтузиастов генетической генеалогии (Веселовского и пр.) удалось выяснить, например, что генофонд древние образцы представителей Ямной культуры в рамках формальных тестов (f3 и D-статистик) наилучшим образом аппроксимируются как результат смешения древнего мезолитического населения севера Европы (в работе их представляют карельские образцы, очевидно из известных захоронений Палеострова) и населения, близкого к современным закавказским популяциям (лучший результат дали армяне из Еревана).
Признаюсь, эти сведения приободрили меня. Дело в том, что последние несколько недель я занимался изучением эволюции аутосомного генофонда беларусов (и своего тоже) из недавно опубликованного набора лаборатории Райха (это одна из усеченных версии их знаменитого кураторского набора Human Origin Dataset).
Как и раньше, для анализа я использовал инструменты разработанные программистами той же лаборатории (Admixtools), а также Alder — программу написанную на основе открытого кода Admixtools, и оптимизированную под более детальный анализ процесса смешивания различных предковых групп.
Так вот, до получения сведений о предварительных результатах попгенетиков, я был немного смущен полученной картиной. У меня получилось вот что. С точки зрения формальной оценки (f3-статистки, аналога более известной p-статистки) лучшие пары адмикса для беларусов (с отрицательным значением Z) представляли собой либо комбинацию мезолитического населения Европы (Loshbour) и современного населения современной Анатолии и ближнего Востока, либо комбинацию ‘генов’ неолитических жителей Европы (LBK380, а также современных сардинцев) и современных америндских популяций (происходящих, как нам известно, из восточной Сибири).
Вот начало списка значимых пар:
Mixe Sardinian Vadim -11.811
Sardinian Mixe Vadim -11.811
Karitiana Sardinian Vadim -11.757
Sardinian Karitiana Vadim -11.757
Zapotec Sardinian Vadim -11.638
Sardinian Zapotec Vadim -11.638
Loschbour Georgian_Megrels Vadim -11.599
Georgian_Megrels Loschbour Vadim -11.599
Piapoco Sardinian Vadim -11.482
Sardinian Piapoco Vadim -11.482
Loschbour Turkish_Trabzon Vadim -11.434
Turkish_Trabzon Loschbour Vadim -11.434
Loschbour Assyrian_WGA Vadim -11.395
Assyrian_WGA Loschbour Vadim -11.395
LBK380 Piapoco Vadim -11.354
Piapoco LBK380 Vadim -11.354
Surui Sardinian Vadim -11.346
Sardinian Surui Vadim -11.346
Loschbour Abkhasian Vadim -11.293
Abkhasian Loschbour Vadim -11.293
Bolivian_LaPaz Sardinian Vadim -11.232
Sardinian Bolivian_LaPaz Vadim -11.232
Loschbour Iranian_Jew Vadim -11.231
Iranian_Jew Loschbour Vadim -11.231
Я выбрал около сотни значимых пар и проверил их достоверность «адмикса) с помощью инструментов D-статистки (qpDstat) в попарном сравнении каждой из значимых комбинаций (начало таблицы):
Vadim Italian_Tuscan : Loschbour Palestinian 0.0293 8.141 best
Vadim Iranian : LBK380 GujaratiC_GIH 0.0245 7.319 best
Vadim Motala12 : Druze Sardinian 0.0125 7.285 best
Vadim Loschbour : Palestinian Albanian 0.0146 7.17 best
Vadim Sardinian : GujaratiC_GIH Iranian 0.0121 7.151 best
Vadim Palestinian : Spanish_Pais_Vasco_IBS GujaratiC_GIH 0.0145 7.126 best
Vadim Egyptian_Comas : Basque_Spanish GujaratiC_GIH 0.0137 7.016 best
Vadim Sardinian : Loschbour Egyptian_Comas 0.0251 6.962 best
Vadim Sardinian : Loschbour Tunisian_Jew 0.0251 6.789 best
Vadim Palestinian : Basque_Spanish GujaratiC_GIH 0.013 6.758 best
Vadim Sardinian : Loschbour Palestinian 0.0237 6.69 best
Vadim Basque_Spanish : Balkar Palestinian 0.0076 6.601 best
Vadim GujaratiC_GIH : Tunisian_Jew Egyptian_Comas 0.0094 6.493 best
Vadim Spanish_Pais_Vasco_IBS : Balkar Palestinian 0.0079 6.458 best
Vadim Loschbour : Druze Italian_WestSicilian 0.0135 6.443 best
Vadim Loschbour : Iranian Albanian 0.0159 6.385 best
Vadim Palestinian : Sardinian Iranian 0.0083 6.344 best
Как видно, лучшая достоверность (обмена генами) у тех пар которые представляют собой комбинацию мезолитических популяций (Loshbour и Motala), популяций Кавказа, южной Европы и центральной Азии.
Это особенно хорошо заметно в тесте f4ratio. Вот например сравнение 2 квадропул, три популяции в каждой из которых идентичны (беларусы, кумыки и Losbour), а четвертая популяция отличается (балкарцы vs. Motala). Результат означает что кроме мезолитического компонента Loshbour (из западной Европы), у беларусов наблюдается эксцесс (28+-0.1%) дополнительного источника мезолитических «генов» (типично для балтийских популяций мезолита вроде Motala)
Vadim Kumyk Loschbour Motala12 : Vadim Kumyk Loschbour Balkar 0.285678 0.096194 2.97
Крайне любопытны и результаты проведенного мной в Alder исследования источников «древного» адмикса у беларусов.
Я выбрал только те пары, в которых амплитуда угасания LD в двух гипотетических популяциях-донорах была сопоставима с амплитудой угасания LD в популяции-реципиенте (т.е у беларусов). Интересно, что только две пары (пенджабцы + Motala) и (иракские евреи + чукчи) дали консистентную попарную подгонку кривой угасания LD с незначительным разбросом амплитуды (15-25%). К слову, комбинация Armenian+Motala-merge (примерно идентичная наиболее устойчивой модели адмикса у жителей ямной культуры) тоже присутствует в списке «успешных» комбинаций, однако кривые угасания LD имеют разную скорость угасания (их амплитуда отличается уже на 55% и поэтому они не консистентны, т.е несовместимы) в попарном режиме сравнения
DATA: success_consistent 0.0042 Belarusian Punjabi_Lahore_PJL Motala_merge 4.49 2.76 2.78 15%
DATA: success_consistent 0.0098 Belarusian Iraqi_Jew Chukchi 4.31 2.2 3.01 25%
DATA: success 0.0065 Belarusian Mongola Motala_merge 4.4 2.64 2.78 28%
DATA: success 0.011 Belarusian Yi Papuan 4.29 2.26 4.66 28%
DATA: success 0.00037 Belarusian Lebanese Papuan 4.98 2.69 4.66 38%
DATA: success 0.041 Belarusian Kusunda Motala_merge 3.98 2.61 2.78 41%
DATA: success 0.013 Belarusian Hezhen Motala_merge 4.25 2.17 2.78 49%
DATA: success 0.037 Belarusian Motala_merge Tu 4.01 2.78 3.13 51%
DATA: success 4.20E-06 Belarusian Kalmyk Motala_merge 5.79 2.36 2.78 54%
DATA: success 0.0086 Belarusian She Motala_merge 4.34 2.58 2.78 54%
DATA: success 0.0019 Belarusian Armenian Motala_merge 4.66 2.14 2.78 55%
DATA: success 0.048 Belarusian Daur Motala_merge 3.94 2.11 2.78 56%
DATA: success 0.0042 Belarusian Motala_merge Miao 4.49 2.78 3.5 59%
DATA: success 0.041 Belarusian Oroqen Motala_merge 3.98 2.28 2.78 59%
DATA: success 0.013 Belarusian Thai Motala_merge 4.25 2.13 2.78 65%
DATA: success 0.043 Belarusian Motala_merge Lahu 3.97 2.78 3.56 71%
DATA: success 0.0049 Belarusian Motala_merge Japanese 4.46 2.78 3.53 72%
Примечательно что для пары Belarusian Armenian Motala_merge Admixtools датирует смешение 114.67+/-20.5 поколений тому назад. А вот датировка адмикса для двух первых пар (последняя колонка это датировка адмикса
Belarusian Punjabi_Lahore_PJL Motala_merge 4.49 2.76 2.78 15% 142.4+/-27.54
Belarusian Iraqi_Jew Chukchi 4.31 2.2 3.01 25% 43.28+/-9.45 То есь самое позднее 3500 лет до нашего времени.Итак, выводы: в эволюционной перспективе, костяк аутосомного генофонда беларусов составляет субстрат мезолитического генетического компонента Европы, к которому примешиваются два потока — один с юга, с наиболее значимым вливанием во времена неолита (земледельцы из Анатолии и ближнего Востока), другой — видимо более поздний (т.к. он отсутствует у ямников) из Сибири.
*Культура боевых топоров, культура шнуровой керамики (нем. Schnurkeramik) — археологическая культура медного и бронзового веков, распространенная на обширных территориях Центральной и Восточной Европы и датированная 3200 г. до н. э./2300 до н. э. — 2300 г. до н. э./1800 г. до н. э. Племена культуры боевых топоров часто считают первыми индоевропейцами на территории Средней Европы
**Я́мная культу́ра (точнее — Древнея́мная культу́рно-истори́ческая о́бщность) — археологическая культура эпохи позднего медного века — раннего бронзового века (3600—2300 до н. э.). Занимала территорию от Южного Приуралья на востоке до Днестра на западе, от Предкавказья на юге до Среднего Поволжья на севере.В рамках ранней версии курганной гипотезы Марии Гимбутас ямная культура связывалась с поздними протоиндоевропейцами.
Кластер древних жителей по своей устойчивости занимает промежуточное место между кластерами древних северных евразийцев и западных европейских охотников-собирателей.
В этот кластер, иерархически близкий популяциям Кавказа и ближнего Востока, предсказуемо входят предстаители самых классических популяции южной Европы — от греков и болгар, до басков и сардинцев. Как уже стало обычным, сардинцы кластеризуются с образцом тирольского человека Этци и женщины из линейноленточной культуры («LBK380»). В большинстве вариантов (2 из трех опубликованных) к этой подгруппе примыкают представители древнейших неолитических культур на территории современной Венгрии — CO1, H4, H3, NE5, NE7). Жители бронзового века (на графике они ошибочно обозначены как Europe оказались посередине между раннеевропейскими охотникам-собирателями и земледельцами.
Добавление к выборке древних геномов «усть-ишимца» и «костенковца» позволило пролить свет на некоторые особенности эволюции популяций центральной и восточной части Евразии. В кластерном анализе (вардовская кластеризация) по 4 первым компонентам PCA усть-ишимец у меня получился в одном кластере с киргизами и кажется селькупами. По первым двум компонентам в том варианте рейховского набора популяций, где нет андаманцев Onge — он попадает в один кластер с австралийскими аборигенами.
Думаю, что onge все же ближе, да к тому же во всех калькуляторах у усть-ишимца максимум «генома» приходится на сочетание южно-индийских и юго-восточноазиатских компонентов. А вот «костенковец» оказывается ближе всего к чувашам и саамам. Что характерно — в предыдущих вариантах, в которых я не использовал костенковца, место костенковца часто занимал AG-2 (Afontova Gora). Также заметна разница между кластерными схемами PC1-2 и PC-1-2-3-4.В первом случае костенковец в одном кластере с индусами, а во-втором с с чувашами и саамами. Характерно, что восточноевразийские палеогеномы Тяньюань и Усть-Ишим входят в один кластер (их положение не сильно меняется), а MA1 нет.

































Для отправки комментария необходимо войти на сайт.