Оглядываясь назад на события и открытия, коими в уходящем 2014 году ознаменовалась область исследований генетики человека, можно смело сказать что уходящий год был годом прорыва в двух принципиально различных направлениях — в палеогенетике, изучающей геномы популяций древних людей прошлого, и в эпигенетике, с помощью которой можно прогнозировать будущее (здоровье и качество жизни) отдельных людей.
Палеогенетика
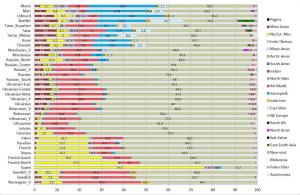
В самом начале 2014 года, на руках немногочисленных исследователей палеогеномов было менее десятка древних геномов человека, опубликованных в предыдущие года. К концу 2014 года опубликован ряд работ, посвященных попыткам генотипирования останков древних людей — от живших несколько тысячелетий назад до «усть-ишимца» с предположительным возрастом около 45 тысяч лет, неандертальцев и «денисовки». Количество таких расшифровок растет все быстрее, что не может не вызывать оптимизма. Вторая половина 2014 года особенно примечательна как количеством подобных публикаций, так и числом полных геномных NGS-сиквенсов древних людей, размещенных в публичных репозиториях (банках геномных данных). Так, в сентябре в Nature была опубликована окончательная версия работы Lazaridis et al. 2014 «Ancient human genomes suggest three ancestral populations for present-day Europeans». Работа получила широкое освещение в СМИ, поскольку аналитическая выборка сэмплов в этом исследовании включала значительное количествао заново генотипированных (на чипе Affymetrix HumanOrigin) образцов ДНК из древних палеолитических стоянок Сибири (Афонтова Гора, Малта), представителя древней индейской культуры Кловис и палеоэскимоса Cаккак. В работе был представлен целый ряд образцов древней ДНК представителей европейских мезолитических и неолитических культур, опубликованных в более ранних работах 2012-2014 годов: Skoglund et a. 2014 «Genomic Diversity and Admixture Differs for Stone-Age Scandinavian Foragers and Farmers»(шведские земледельцы и охотники собиратели эпохи неолита); Olalde et al. 2014 «Derived immune and ancestral pigmentation alleles in a 7,000-year-old Mesolithic European» (дДНК мезолитического населения Иберийского полуострова) и т.д.
Опубликованные геномы так и остались бы достоянием небольшой группы ученых, и по-прежнему бы использовались бы только для сравнения с абстрактными и анонимизрованных данными референсных популяций человека, если бы усилиями пары любителей (прежде всего усилиям Чандракумара) палеогеномы не были преобразованы в привычные и удобные для популяционного анализа форматы BAM, VCF и Plink binary, а также в стандартный формат геномных данных от FTDNA. По своей сути, преобразование состояло в сложной процедуре сборки генома из библиотек коротких геномных ридов (в формате sra., в котором эти риды хранятся в репозиториях крупных баз геномных данных). Полученные сборки геномов в формате sam/bam cравниваются с референсным геномом человека, и отличающиеся одиночные нуклеотидные полиморфизмы сохраняются в VCF файл. Здесь нужно помнить о том, что в этой процедуре не учитывался параметр качества сиквенса PHRED score. Традиционно рекомендуется использовать только те базовые пары, PHRED score которых превышает 30, т.е чья точность определения составляет 99.9% (или 1 ошибка на 1000 базовых пар). Кроме того, в этой процедуре разработчик не учел влияние постмортальных изменений ДНК. Cледует помнить, что ДНК, как и любая биомолекула, способна вступать в химические реакции с окружающим миром, тут-то и появляются различные модификации нуклеотидов (особенно по краям фрагментов древней ДНК). Наиболее частая постмортальная мутация — дезаминирование цитозинов (C), приводящая к возникновению урацилов (U) в последовательности древней ДНК, которые при проведении ПЦР многократно копируются «бездушным» ферментом ДНК-полимеразой как тимин (Т). Именно по этой причине, при оценке достоверности снипов в полученных из палеогеномов вариантах особое внимание требуется уделять транзициям C->T и G->A. Если при подсчете вариантов окажется, что такие транзиции встречаются чаще ожидаемого, то можно сделать вывод о существенном повреждении палео-ДНК. И хотя по причине игнорирования этих ограничений, автором было получено большое количество снипов, в некоторых случаях, например при объединении полученных данных NGS c данными генотипирования с помощью классических технологий миркочипов, использование таких данных может существенно уменьшить качество интерпретации.
Тем не менее, благодаря этим усилиям, и не в меньшей степени, благодаря соотрудничеству с порталом Gedmatch и компанией FTDNA, большинство клиентов ведущих компаний на рынке персональной геномики и генетической генеалогии (таких как 23andme, и FTDNA) могут сравнить свои данные с данными древних геномов либо путем сравнения частото аллелей, либо посегментно сравнивая свои хромосомы с гомологичными хромосомами древних геномов. Более того, Феликс Чандракумар пошел дальше и разместил 10 наиболее качественных палеогеномов (т.е палеогеномов с наибольшим числом перекрывающихся разными микроматрицами снипов) на FTDNA. Таким образом, с помощью сервиса MyOrigins FTDNA, исследователи могут установить распределение «этно-популяционных составляющих» или «предковых этнопопуляционных компонентов» в этих древних геномов. Нужно помнить, конечно же, что в случае с наиболее древними геномами (геномами неандертальца, усть-ишимца и т.д.) полученное распределение более молодых компонентов (полученных из современных популяций) нельзя интерпретировать буквально.










Эту замечательную функцию дополняют калькуляторы Eurogenes, благодаря которым любой интересующийся человек может посмотреть, какой процент его/ее генома приходится на тот или иной древний геном. Для людей, озабоченных вопросами анонимности, Феликс разработал отдельное десктопное приложение — калькулятор древней ДНК. Этот калькулятор показывает, какой процент ДНК (составных сегментов) аутосомной ДНК клиента попадает в каждый из 30 образцов древней ДНК . Другими словами, он показывает процент общих предков в сравнении современного ДНК и палеоДНК.
Подводя итоги года, можно сказать, что в области изучения древней ДНК все ожидания были оправданы.
Эпигенетика
Под эпигенетикой обычно понимают область знаний о совокупности свойств организма, которые не закодированы непосредственно в геноме, но могут и должны передаваться по наследству.Эпигенетика может быть определена как изучение механизмов контроля активности генов во времени и пространстве в процессе развития сложных организмов. К настоящему времени обнаружены и описаны различные механизмы контроля активности генов, однако в уходящем 2014 году особое внимание ученые уделяли изучению одного из таких механизмов — ферментативному (энзиматическому) метилированию самой генетической матрицы, то есть ДНК.
Метилирование — это изменение молекулы ДНК путем присоединения метильной группы (-СH3) к нуклеотиду C, причем необходимо, чтобы за С следовал нуклеотид G. Последовательность нуклеотидов -CG- называется СpG динуклеотидом, или CpG сайтом. Метилирование происходит не во всех клетках одновременно, поэтому говорят о проценте метилирования определенного CpG сайта.метилирование ДНК ощутимо сказывается на её взаимодействии (связывании) с различными белками. Во многих случаях метилирование по цитозиновым остаткам препятствует связыванию специфично реагирующих с ДНК ядерных белков (факторов), которые, собственно, и осуществляют разные генетические процессы, в том числе транскрипцию, репликацию и репарацию.Как известно, метилирование играет важнейшую роль в механизме экспрессии (т.е качественном и количественном проявлени) генотипа в фенотип. оказано, что с изменением профиля метилирования связаны такие заболевания, как различные виды рака, диабет первого и второго рода, шизофрения и т.д. Поэтому важно уметь анализировать профиль метилирования генома, и здесь перед энзимологией расскрываются огромные перспективы. Например, в 2014 году компания «СибЭнзайм» открыла новый фермент, на базе которого разработали новый метод детекции. Он позволяет определять, включен или выключен интересующий вас ген — э то управляющий механизм в организме, именно отключение отдельных генов ученые связывают с развитием рака:

С технической точки зрения, изучение метиляции ДНК происходит с использованием модифицированного варианта ChiPSeq (это комбинированный вариант иммунопреципитации хроматина (ChIP) и высокоэффективного секвенирования ДНК для определения участков связывания ДНК и белков). Не вдаваясь в биолого-химические подробности этого модифицированного метода, его можно кратко описать следующим образом. Каждый CpG сайт измеряется с помощью двух флуоресцентных проб. Флуоресцентный сигнал проб пропорционален соответственно количеству метилированных и неметилированных CpG сайтов в тестируемом образце. Полученные данные образуют собой профиль метилирования, который удобно сравнивать с различными референсными образцами. Как уже говорилось выше, этот профиль можно использовать не только для медицинских целей (например, для изучения эпигенетических факторов развития различных заболеваний), но и для более общих целей. В недавном исследовании, проведённом специалистами из Калифорнийского университета (UCLA), выявило биологические часы, встроенные в геном человека и оно впервые определило, что внутренние часы в состоянии точно оценить возраст различных человеческих органов, тканей и клеток. Исследователи обратили свое внимание на метилировании – естественном процессе, изменяющем химический состав ДНК. Он изучил 121 набор данных, собранных ранее исследователями, изучавшими метилирование здоровых и раковых тканей человека. Проанализировав информацию по 8000 образцов из 51 типа тканей и клеток со всего тела, исследователи смогли определить, как возраст влияет на уровни метилирования с рождения до 101 года. Он определил, что метилирование работает на 353 участках ДНК, которые изменяются с возрастом. Таким образом, профиль метилирования ДНК представляет собой наиболее надежную метрику для расчетов биологического возраста как отдельных органов, так и всего организма.
Принимая это во внимание, можно сказать что и в последующие года эпигенетику ожидают радужные перспективы.


















Для отправки комментария необходимо войти на сайт.